Julia でロジスティック回帰 - glm
前回 に続き、今回も Julia で GLM を実施します。
今回は 「R でロジスティック回帰 - glm, MCMCpack」 のロジスティック回帰(GLM)を Julia で実装してみました。
サンプルソースは http://github.com/fits/try_samples/tree/master/blog/20150309/
はじめに
GLM 等のパッケージは 前回 と同じものを使用します。
データは R で試した時のものをそのまま使います。(「R でロジスティック回帰 - glm, MCMCpack」 参照)
データ data4a.csv
N,y,x,f 8,1,9.76,C 8,6,10.48,C 8,5,10.83,C ・・・
データの内容は以下の通りで、個体 i それぞれにおいて 「 個の観察種子のうち生きていて発芽能力があるものは
個」 となっています。
| 項目 | 内容 |
|---|---|
| N | 観察種子数 |
| y | 生存種子数 |
| x | 植物の体サイズ |
| f | 施肥処理 (C: 肥料なし, T: 肥料あり) |
体サイズ x と肥料による施肥処理 f が種子の生存する確率(ある個体 i から得られた種子が生存している確率)にどのように影響しているかをロジスティック回帰で解析します。
GLM によるロジスティック回帰
Julia の GLM パッケージを使う場合の注意点は下記の通りです。
- (1) Binomial (二項分布) を使う場合は応答変数(下記の y に該当)の値が 0.0 ~ 1.0 内でなければならない
- (2) readtable ではカテゴリ型変数 (R の因子型に該当) 化を実施しない
(1) への対応として、y を N で割った値 yn を応答変数(目的変数)として使いました。
今のところ d[:yn] = d[:y] / d[:N] とは書けないようなので map 関数を使っています。
R の read.csv 関数等では f のような項目は因子型になると思いますが、Julia の readtable では (2) のようにカテゴリ型変数とはならず単なる文字列の配列となります。 (d[:f] の型は DataArrays.DataArray{UTF8String,1} になる)
glm 関数で文字列の変数は使えないので、カテゴリ型変数への変換が必要となります。
DataFrames パッケージにはカテゴリ型への変換関数 pool が用意されているので、下記では f 項目の値を pool 関数で変換し ff へ設定しています。 (d[:ff] の型は DataArrays.PooledDataArray{UTF8String,UInt8,1} になる)
logisticGlm.jl
using DataFrames, GLM d = readtable("data4a.csv") # (1) 生存率 y / N の算出 d[:yn] = map(x -> d[:y][x] / d[:N][x], 1:nrow(d)) # (2) カテゴリ型へ変換 (DataArrays.PooledDataArray{UTF8String,UInt8,1} になる) d[:ff] = pool(d[:f]) # 以下でも可 (ただし、DataArrays.PooledDataArray{UTF8String,UInt32,1} になる) # d[:ff] = convert(PooledDataArray, d[:f]) res = glm(yn~x + ff, d, Binomial()) println(res)
実行結果は以下のようになりました。 (R で実施したときと同じ Estimate 値)
実行結果
> julia logisticGlm.jl
DataFrames.DataFrameRegressionModel{GLM.GeneralizedLinearModel{GLM.GlmResp{Array{Float64,1},Distributions.Binomial,GLM.LogitLink},GLM.DensePredChol{Float64}},Float64}:
Coefficients:
Estimate Std.Error z value Pr(>|z|)
(Intercept) -19.5361 3.99861 -4.88572 <1e-5
x 1.95241 0.392777 4.97077 <1e-6
ff - T 2.02151 0.654152 3.09027 0.0020
ちなみに、d[:f] の内容と pool・convert 関数の適用結果は以下のようになります。
julia> d[:f]
100-element DataArrays.DataArray{UTF8String,1}:
"C"
"C"
・・・
"T"
"T"
julia> pool(d[:f])
100-element DataArrays.PooledDataArray{UTF8String,UInt8,1}:
"C"
"C"
・・・
"T"
"T"
julia> convert(PooledDataArray, d[:f])
100-element DataArrays.PooledDataArray{UTF8String,UInt32,1}:
"C"
"C"
・・・
"T"
"T"
PooledDataArray
PooledDataArray の水準名の一覧は levels 関数か pool フィールドを使って取得できます。
julia> levels(d[:ff])
2-element Array{UTF8String,1}:
"C"
"T"
julia> d[:ff].pool
2-element Array{UTF8String,1}:
"C"
"T"
水準名と数値のマッピングは levelsmap 関数で取得できます。
julia> levelsmap(d[:ff])
Dict{UTF8String,Int64} with 2 entries:
"T" => 2
"C" => 1
また、refs フィールドで各データに割り当てられた数値を取得できます。
julia> d[:ff].refs
100-element Array{UInt8,1}:
0x01
0x01
・・・
0x02
0x02
予測線の描画
次に glm の結果 (DataFrameRegressionModel) を使って予測腺を描画します。
glm の結果では、「肥料あり "T" => 1、肥料なし "C" => 0」 の扱いになっているので、predict する際に肥料ありは 1 を肥料なしは 0 の配列を渡します。
predict の結果は生存率 yn の予測値なので、グラフへ描画する際に観察種子数 N の最大値 (今回は全て 8 なので最大値とする必要はない) を乗算しています。
なお、rep 関数を使うと指定の配列を指定回数繰り返した配列を作成できます。
logisticGlm_draw.jl
using DataFrames, GLM, Gadfly ・・・ res = glm(yn~x + ff, d, Binomial()) # x の最小値 7.66 ~ 最大値 12.44 まで 0.1 刻みのデータを用意 xx = [minimum(d[:x]):0.1:maximum(d[:x])] # 肥料あり "T" の予測値を算出 rt = predict(res, DataFrame(n = rep([1], length(xx)), x = xx, ff = rep([1], length(xx)))) # 肥料なし "C" の予測値を算出 rc = predict(res, DataFrame(n = rep([1], length(xx)), x = xx, ff = rep([0], length(xx)))) p = plot( layer(d, x = "x", y = "y", color = "f", Geom.point), # 肥料あり "T" の予測線を赤で描画 layer(x = xx, y = maximum(d[:N]) * rt, Geom.line, Theme(default_color = color("red"))), # 肥料なし "C" の予測線を緑で描画 layer(x = xx, y = maximum(d[:N]) * rc, Geom.line, Theme(default_color = color("green"))) ) draw(PNG("logisticGlm_draw.png", 500px, 400px), p)
実行結果
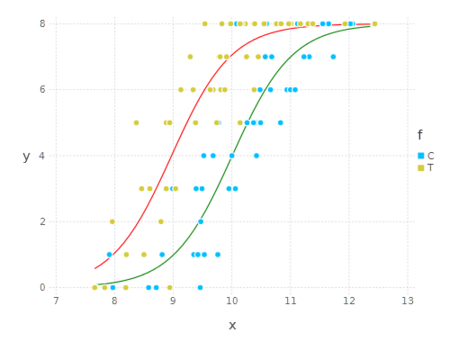
なお、rt と rc の算出処理を PooledDataArray を使って実装すると以下のようになります。
PooledDataArray では "T" => 2, "C" => 1 となっているため、"T" => 1, "C" => 0 でそれぞれ predict するように <PooledDataArray変数>.refs - 1 としています。
・・・ xx = [minimum(d[:x]):0.1:maximum(d[:x])] # 肥料あり "T" のカテゴリ型データを作成 (d[:ff] の水準を使用) ft = PooledDataArray(rep([utf8("T")], length(xx)), d[:ff].pool) # 肥料なし "C" のカテゴリ型データを作成 (d[:ff] の水準を使用) fc = PooledDataArray(rep([utf8("C")], length(xx)), d[:ff].pool) # 肥料あり "T" の予測値を算出 rt = predict(res, DataFrame(n = rep([1], length(xx)), x = xx, ff = ft.refs - 1)) # 肥料なし "C" の予測値を算出 rc = predict(res, DataFrame(n = rep([1], length(xx)), x = xx, ff = fc.refs - 1)) ・・・