Word2Vec を用いた併売の分析 - gensim
「トピックモデルを用いた併売の分析」ではトピックモデルによる併売の分析を試しましたが、今回は gensim の Word2Vec で試してみました。
ソースは http://github.com/fits/try_samples/tree/master/blog/20180617/
はじめに
データセット
これまで は適当に作ったデータセットを使っていましたが、今回は R の Groceries データセット ※ をスペース区切りのテキストファイル(groceries.txt)にして使います。(商品名にスペースを含む場合は代わりに _ を使っています)
※ ある食料雑貨店における 30日間の POS データ
groceries.txt
citrus_fruit semi-finished_bread margarine ready_soups tropical_fruit yogurt coffee whole_milk pip_fruit yogurt cream_cheese_ meat_spreads other_vegetables whole_milk condensed_milk long_life_bakery_product whole_milk butter yogurt rice abrasive_cleaner rolls/buns other_vegetables UHT-milk rolls/buns bottled_beer liquor_(appetizer) pot_plants whole_milk cereals ・・・ cooking_chocolate chicken citrus_fruit other_vegetables butter yogurt frozen_dessert domestic_eggs rolls/buns rum cling_film/bags semi-finished_bread bottled_water soda bottled_beer chicken tropical_fruit other_vegetables vinegar shopping_bags
R によるアソシエーションルールの抽出結果
参考のため、まずは R を使って Groceries データセットを apriori で処理しました。
リフト値を優先するため、支持度 (supp) と確信度 (conf) を低めの値にしています。
groceries_apriori.R
library(arules) data(Groceries) params <- list(supp = 0.001, conf = 0.1) rules <- apriori(Groceries, parameter = params) inspect(head(sort(rules, by = "lift"), 10))
実行結果
> Rscript groceries_apriori.R
・・・
lhs rhs support confidence lift count
[1] {bottled beer,
red/blush wine} => {liquor} 0.001931876 0.3958333 35.71579 19
[2] {hamburger meat,
soda} => {Instant food products} 0.001220132 0.2105263 26.20919 12
[3] {ham,
white bread} => {processed cheese} 0.001931876 0.3800000 22.92822 19
[4] {root vegetables,
other vegetables,
whole milk,
yogurt} => {rice} 0.001321810 0.1688312 22.13939 13
[5] {bottled beer,
liquor} => {red/blush wine} 0.001931876 0.4130435 21.49356 19
[6] {Instant food products,
soda} => {hamburger meat} 0.001220132 0.6315789 18.99565 12
[7] {curd,
sugar} => {flour} 0.001118454 0.3235294 18.60767 11
[8] {soda,
salty snack} => {popcorn} 0.001220132 0.1304348 18.06797 12
[9] {sugar,
baking powder} => {flour} 0.001016777 0.3125000 17.97332 10
[10] {processed cheese,
white bread} => {ham} 0.001931876 0.4634146 17.80345 19
bottled beer と red/blush wine で liquor が同時に買われやすい、hamburger meat と soda で Instant food products が同時に買われやすいという結果(アソシエーションルール)が出ています。
Word2Vec の適用
それでは groceries.txt を gensim の Word2Vec で処理してみます。
とりあえず iter を 500 に min_count を 1 にしてみました。
なお、購入品目の多い POS データを処理する場合は window パラメータを大きめにすべきかもしれません。(今回はデフォルト値の 5)
今回は Jupyter Notebook で実行しています。
Word2Vec モデルの構築
from gensim.models import word2vec sentences = word2vec.LineSentence('groceries.txt') model = word2vec.Word2Vec(sentences, iter = 500, min_count = 1)
類似品の算出
まずは、wv.most_similar で類似単語(商品)を抽出してみます。
pork の類似単語
model.wv.most_similar('pork')
[('turkey', 0.5547687411308289),
('ham', 0.49448296427726746),
('pip_fruit', 0.46879759430885315),
('tropical_fruit', 0.4383287727832794),
('butter', 0.43373265862464905),
('frankfurter', 0.4334157109260559),
('root_vegetables', 0.4249211549758911),
('citrus_fruit', 0.4246293306350708),
('chicken', 0.42378148436546326),
('sausage', 0.41153857111930847)]
微妙なものも含んでいますが、それなりの結果になっているような気もします。
most_similar はベクトル的に類似している単語を抽出するため、POS データを処理する場合は競合や代用品の抽出に使えるのではないかと思います。
併売の分析
併売の商品はお互いに類似していないと思うので most_similar は役立ちそうにありませんが、それでも何らかの関係性はありそうな気がします。
そこで、指定した単語群の中心となる単語を抽出する predict_output_word を使えないかと思い、R で抽出したアソシエーションルールの組み合わせで試してみました。
predict_output_word の検証
bottled_beer と red/blush_wine
model.predict_output_word(['bottled_beer', 'red/blush_wine'])
[('liquor', 0.22384332),
('prosecco', 0.04933687),
('sparkling_wine', 0.0345262),
・・・]
R の結果に出てた liquor が先頭(確率が最大)に来ています。
bottled_beer と red/blush_wine
model.predict_output_word(['hamburger_meat', 'soda'])
[('Instant_food_products', 0.054281656),
('canned_vegetables', 0.029985178),
('pasta', 0.025487985),
・・・]
ここでも R の結果に出てた Instant_food_products が先頭に来ています。
ham と white_bread
model.predict_output_word(['ham', 'white_bread'])
[('processed_cheese', 0.20990367),
('sweet_spreads', 0.024131883),
('spread_cheese', 0.023222428),
・・・]
こちらも同様です。
root_vegetables と other_vegetables と whole_milk と yogurt
model.predict_output_word(['root_vegetables', 'other_vegetables', 'whole_milk', 'yogurt'])
[('herbs', 0.024541182),
('liver_loaf', 0.019327056),
('turkey', 0.01775743),
('onions', 0.01760579),
('specialty_cheese', 0.014991459),
('packaged_fruit/vegetables', 0.014529809),
('spread_cheese', 0.012931713),
('meat', 0.012434797),
('beef', 0.011924307),
('butter_milk', 0.011828974)]
R の結果にあった rice はこの中には含まれていません。
curd と sugar
model.predict_output_word(['curd', 'sugar'])
[('flour', 0.076272935),
('pudding_powder', 0.055790607),
('baking_powder', 0.026003197),
・・・]
R の結果に出てた flour (小麦粉) が先頭に来ています。
soda と salty_snack
model.predict_output_word(['soda', 'salty_snack'])
[('popcorn', 0.05830234),
('nut_snack', 0.046429735),
('chewing_gum', 0.0213278),
・・・]
こちらも同様です。
sugar と baking_powder
model.predict_output_word(['sugar', 'baking_powder'])
[('flour', 0.11954326),
('cooking_chocolate', 0.046284538),
('pudding_powder', 0.03714784),
・・・]
こちらも同様です。
以上のように、少なくとも 2品を指定した場合の predict_output_word の結果は R で抽出したアソシエーションルールに合致しているようです。
Word2Vec のパラメータに左右されるのかもしれませんが、この結果を見る限りでは predict_output_word を 3品の併売の組み合わせ抽出に使えるかもしれない事が分かりました。
3品の併売
次に predict_output_word で 2品に対する 1品を確率の高い順に抽出してみました。
なお、ここでは 3品の組み合わせの購入数が 10 未満のものは除外するようにしています。
from collections import Counter import itertools # 3品の組み合わせのカウント tri_counter = Counter([c for ws in sentences for c in itertools.combinations(sorted(ws), 3)]) # 2品の組み合わせを作成 pairs = itertools.combinations(model.wv.vocab.keys(), 2) sorted([ (p, item, prob) for p in pairs for item, prob in model.predict_output_word(p) if prob >= 0.05 and tri_counter[tuple(sorted([p[0], p[1], item]))] >= 10 ], key = lambda x: -x[2])
[(('bottled_beer', 'red/blush_wine'), 'liquor', 0.22384332),
(('white_bread', 'ham'), 'processed_cheese', 0.20990367),
(('bottled_beer', 'liquor'), 'red/blush_wine', 0.16274776),
(('sugar', 'baking_powder'), 'flour', 0.11954326),
(('curd', 'sugar'), 'flour', 0.076272935),
(('margarine', 'sugar'), 'flour', 0.07422828),
(('flour', 'sugar'), 'baking_powder', 0.07345509),
(('sugar', 'whipped/sour_cream'), 'flour', 0.072731614),
(('rolls/buns', 'hamburger_meat'), 'Instant_food_products', 0.06818052),
(('sugar', 'root_vegetables'), 'flour', 0.0641469),
(('tropical_fruit', 'white_bread'), 'processed_cheese', 0.061861355),
(('soda', 'ham'), 'processed_cheese', 0.06138085),
(('white_bread', 'processed_cheese'), 'ham', 0.061199907),
(('whole_milk', 'ham'), 'processed_cheese', 0.059773713),
(('beef', 'root_vegetables'), 'herbs', 0.059243686),
(('sugar', 'whipped/sour_cream'), 'baking_powder', 0.05871357),
(('soda', 'salty_snack'), 'popcorn', 0.05830234),
(('soda', 'popcorn'), 'salty_snack', 0.05819882),
(('red/blush_wine', 'liquor'), 'bottled_beer', 0.057226427),
(('flour', 'baking_powder'), 'sugar', 0.05517209),
(('soda', 'hamburger_meat'), 'Instant_food_products', 0.054281656),
(('processed_cheese', 'ham'), 'white_bread', 0.053193364),
(('other_vegetables', 'ham'), 'processed_cheese', 0.052585844)]
R で抽出したアソシエーションルールと同じ様な結果が出ており、それなりの結果が出ているように思います。
skip-gram の場合
gensim の Word2Vec はデフォルトで CBoW を使うようですので、skip-gram の場合にどうなるかも簡単に確認してみました。
skip-gram の使用
model = word2vec.Word2Vec(sentences, iter = 500, min_count = 1, sg = 1)
まずは predict_output_word の結果をいくつか見てみます。
先頭(確率が最大のもの)は変わらないようですが、CBoW よりも確率の値が全体的に低くなっているようです。
bottled_beer と red/blush_wine
model.predict_output_word(['bottled_beer', 'red/blush_wine'])
[('liquor', 0.076620705),
('prosecco', 0.030791236),
('liquor_(appetizer)', 0.027123762),
・・・]
hamburger_meat と soda
model.predict_output_word(['hamburger_meat', 'soda'])
[('Instant_food_products', 0.022627866),
('pasta', 0.018009944),
('canned_vegetables', 0.01685342),
・・・]
root_vegetables と other_vegetables と whole_milk と yogurt
model.predict_output_word(['root_vegetables', 'other_vegetables', 'whole_milk', 'yogurt'])
[('herbs', 0.015105391),
('turkey', 0.014365919),
('rice', 0.01316431),
・・・]
ここでは、CBoW で 10番以内に入っていなかった rice が入っています。
次に、先程と同様に predict_output_word で 3品の組み合わせを確率順に抽出してみます。
確率の値が全体的に下がっているため、最小値の条件を 0.02 へ変えています。
predict_output_word を使った 3品の組み合わせ抽出
・・・ sorted([ (p, item, prob) for p in pairs for item, prob in model.predict_output_word(p) if prob >= 0.02 and tri_counter[tuple(sorted([p[0], p[1], item]))] >= 10 ], key = lambda x: -x[2])
[(('bottled_beer', 'red/blush_wine'), 'liquor', 0.076620705),
(('bottled_beer', 'liquor'), 'red/blush_wine', 0.0712179),
(('white_bread', 'ham'), 'processed_cheese', 0.039820198),
(('red/blush_wine', 'liquor'), 'bottled_beer', 0.031292748),
(('sugar', 'baking_powder'), 'flour', 0.030803043),
(('sugar', 'whipped/sour_cream'), 'flour', 0.029322423),
(('margarine', 'sugar'), 'flour', 0.027827),
(('beef', 'root_vegetables'), 'herbs', 0.02740662),
(('curd', 'sugar'), 'flour', 0.025570681),
(('flour', 'sugar'), 'baking_powder', 0.025403246),
(('tropical_fruit', 'root_vegetables'), 'turkey', 0.025329975),
(('whole_milk', 'ham'), 'processed_cheese', 0.024535457),
(('rolls/buns', 'hamburger_meat'), 'Instant_food_products', 0.02427808),
(('flour', 'baking_powder'), 'sugar', 0.023779714),
(('tropical_fruit', 'white_bread'), 'processed_cheese', 0.023528077),
(('sugar', 'root_vegetables'), 'flour', 0.023394365),
(('soda', 'salty_snack'), 'popcorn', 0.02322538),
(('whole_milk', 'sugar'), 'flour', 0.023202542),
(('fruit/vegetable_juice', 'ham'), 'processed_cheese', 0.023127634),
(('butter', 'root_vegetables'), 'herbs', 0.02304014),
(('soda', 'ham'), 'processed_cheese', 0.022633638),
(('soda', 'hamburger_meat'), 'Instant_food_products', 0.022627866),
(('citrus_fruit', 'sugar'), 'flour', 0.022040429),
(('bottled_beer', 'soda'), 'liquor', 0.02189085),
(('processed_cheese', 'ham'), 'white_bread', 0.021692872),
(('yogurt', 'sugar'), 'flour', 0.021522585),
(('tropical_fruit', 'other_vegetables'), 'turkey', 0.021456005),
(('other_vegetables', 'beef'), 'herbs', 0.021407435),
(('white_bread', 'processed_cheese'), 'ham', 0.021362728),
(('curd', 'root_vegetables'), 'herbs', 0.021005861),
(('other_vegetables', 'ham'), 'processed_cheese', 0.020965746),
(('root_vegetables', 'whipped/sour_cream'), 'herbs', 0.020788824),
(('other_vegetables', 'root_vegetables'), 'herbs', 0.020782541),
(('sugar', 'whipped/sour_cream'), 'baking_powder', 0.02058014),
(('whole_milk', 'sugar'), 'rice', 0.020371588),
(('root_vegetables', 'frozen_vegetables'), 'herbs', 0.02027719),
(('whole_milk', 'Instant_food_products'), 'hamburger_meat', 0.020258738),
(('citrus_fruit', 'root_vegetables'), 'herbs', 0.020241175)]
最小値の条件を下げたために、より多くの組み合わせを抽出していますが、CBoW の結果と大きな違いは無さそうです。
トピックモデルを用いた併売の分析 - gensim の LdaModel 使用
トピックモデルは潜在的なトピックから文書中の単語が生成されると仮定するモデルのようです。
であれば、これを「Python でアソシエーション分析」で行ったような併売の分析に適用するとどうなるのか気になったので、gensim の LdaModel を使って同様のデータセットを LDA(潜在的ディリクレ配分法)で処理してみました。
ソースは http://github.com/fits/try_samples/tree/master/blog/20180313/
1. はじめに
データセット
gensim で LDA を処理する場合、通常は以下のような lowcorpus フォーマットを使った方が簡単なようです。(LowCorpus で処理できるので)
<文書数> <文書1の単語1> <文書1の単語2> ・・・ <文書2の単語1> <文書2の単語2> ・・・ ・・・
ただ、1行目が冗長なように思うので、今回は word2vec 等でも使えるように 1行目を除いたデータファイルを使います。
内容としては 「Python でアソシエーション分析」 の data.basket ファイルをスペース区切りにしただけです。
data.txt
C S M R T Y C P Y C M O W L R O U R L P W C T O W B C C T W B Y F D ・・・ R P S B B S B F C F N
2. LDA の適用
(1) 辞書とコーパスの作成
まずは、ファイルを読み込んで辞書 Dictionary とコーパスを作成します。
単語部分が文字列のままでは処理できないため、単語を一意の ID(数値)へマッピングする Dictionary を用意し、doc2bow で文書を [(<単語ID>, <出現数>), ・・・] のような形式 bag-of-words へ変換しコーパスを作ります。
word2vec.LineSentence を用いてデータファイルを読み込み、併売の分析という点から単一要素の行を除外してみました。
from gensim.corpora import Dictionary from gensim.models import word2vec # 単一要素の行は除外 sentences = [s for s in word2vec.LineSentence('data.txt') if len(s) >= 2] dic = Dictionary(sentences) corpus = [dic.doc2bow(s) for s in sentences]
変数の内容はそれぞれ以下のようになります。
sentences の内容
[['C', 'S', 'M', 'R'], ['T', 'Y', 'C'], ['P', 'Y', 'C', 'M'], ・・・]
dic の内容
{0: 'C',
1: 'M',
2: 'R',
3: 'S',
・・・
16: 'A',
17: 'G',
18: 'Z'}
corpus の内容
[[(0, 1), (1, 1), (2, 1), (3, 1)], [(0, 1), (4, 1), (5, 1)], [(0, 1), (1, 1), (5, 1), (6, 1)], ・・・]
(2) LdaModel 作成
LdaModel は (1) の辞書とコーパスを使って作成できます。
id2word は必須ではありませんが、使用するメソッド次第で必要になるようです。
random_state を指定しない場合、ランダムな値が適用され実行の度に結果が異なります。
from gensim.models.ldamodel import LdaModel lda = LdaModel(corpus = corpus, id2word = dic, num_topics = 8, alpha = 0.01, random_state = 1)
num_topics と alpha
ここで、トピック数 num_topics と alpha の値が重要となります。
トピックが多すぎると、どの文書にも該当しない(該当する確率が非常に低い)無駄なトピックが作られてしまいますし、逆に少なすぎるとあまり特徴の無いトピックが出来て有用な結果が得られないかもしれません。
alpha はデフォルトで 1 / num_topics の値を適用するようになっていますが、alpha の値によって文書あたりの該当トピック数が大きく変化するため注意が必要です。(大きいとトピック数が増えます)
それでは、alpha の値による影響を確認してみます。
LdaModelオブジェクト[bow] か get_document_topics(bow) を用いると、文書(bag-of-words) に対して確率が 0.01 (デフォルト値)以上のトピックを取得でき、内容は [(トピックID, 値), (トピックID, 値), ・・・] となっています。
from statistics import mean for t in range(4, 21, 4): for n in range(1, 10, 2): a = n / 100 lda = LdaModel(corpus = corpus, id2word = dic, num_topics = t, alpha = a, random_state = 1) # 文書の平均トピック数を算出 r = mean([len(lda[c]) for c in corpus]) print(f"num_topics = {t}, alpha = {a}, mean = {r}")
結果は以下のようになりました。
併売の分析として考えると、併売されやすいものは同じトピックに集まって欲しいので、平均トピック数の少ない方が望ましいと考えられます。
以下の中では num_topics = 8, alpha = 0.01 が良さそうですので、以後はこの値を使って処理する事にします。
num_topics, alpha と文書あたりの平均トピック数
num_topics = 4, alpha = 0.01, mean = 1.0675675675675675 num_topics = 4, alpha = 0.03, mean = 1.9054054054054055 num_topics = 4, alpha = 0.05, mean = 3.3378378378378377 num_topics = 4, alpha = 0.07, mean = 3.8378378378378377 num_topics = 4, alpha = 0.09, mean = 3.9594594594594597 num_topics = 8, alpha = 0.01, mean = 1.0405405405405406 num_topics = 8, alpha = 0.03, mean = 3.0405405405405403 num_topics = 8, alpha = 0.05, mean = 6.418918918918919 num_topics = 8, alpha = 0.07, mean = 7.648648648648648 num_topics = 8, alpha = 0.09, mean = 7.905405405405405 num_topics = 12, alpha = 0.01, mean = 1.054054054054054 num_topics = 12, alpha = 0.03, mean = 4.202702702702703 num_topics = 12, alpha = 0.05, mean = 9.486486486486486 num_topics = 12, alpha = 0.07, mean = 11.41891891891892 num_topics = 12, alpha = 0.09, mean = 11.85135135135135 num_topics = 16, alpha = 0.01, mean = 1.1081081081081081 num_topics = 16, alpha = 0.03, mean = 5.351351351351352 num_topics = 16, alpha = 0.05, mean = 12.594594594594595 num_topics = 16, alpha = 0.07, mean = 14.432432432432432 num_topics = 16, alpha = 0.09, mean = 15.81081081081081 num_topics = 20, alpha = 0.01, mean = 1.1081081081081081 num_topics = 20, alpha = 0.03, mean = 6.527027027027027 num_topics = 20, alpha = 0.05, mean = 13.297297297297296 num_topics = 20, alpha = 0.07, mean = 17.972972972972972 num_topics = 20, alpha = 0.09, mean = 19.743243243243242
ちなみに、トピック数 20 で処理すると、どの文書にも(0.01 以上で)該当しないトピックが 4個程度発生しました。そのため、このデータセットでは 16 程度がトピック数の最大値だと思われます。
また、参考のために主成分分析(PCA)で処理してみると、次元数 8 の場合に寄与率の累計が 0.76、16 の場合に 0.98 となりました。
coherence と perplexity
一般的なトピックモデルの評価指標としては coherence (トピック性能) と perplexity (予測性能) というものがあるようです。
通常、perplexity は学習用と評価用にデータを分けて算出するようですが、とりあえず今回はデータを分けずに算出してみました。
coherence の算出方法として u_mass 以外にも c_v や c_uci 等が用意されていましたが、LdaModel の top_topics メソッドのデフォルトが u_mass を使っていたのでこれに倣いました。
ソース ldamodel.py を見たところ、log_perplexity の戻り値は単語あたりの bound (perwordbound) となっており、perplexity は 2 の -perwordbound 乗 で算出するようでした。
import numpy as np from gensim.models.ldamodel import CoherenceModel for i in range(1, 31): lda = LdaModel(corpus = corpus, id2word = dic, num_topics = i, alpha = 0.01, random_state = 1) cm = CoherenceModel(model = lda, corpus = corpus, coherence = 'u_mass') coherence = cm.get_coherence() perwordbound = lda.log_perplexity(corpus) perplexity = np.exp2(-perwordbound) print(f"num_topics = {i}, coherence = {coherence}, perplexity = {perplexity}")
結果は以下の通りです。
coherence は大きい方が望ましく perplexity は小さい方が望ましいのだと思うのですが、単純にトピック数の大小に影響されているこの結果を見る限りは、今回の用途には適していないように思います。(もしくは、算出方法に誤りがあるのかも)
coherence と perplexity 結果
topic_num = 1, coherence = -10.640923461890344, perplexity = 6.681504324473536 topic_num = 2, coherence = -10.564527218339581, perplexity = 7.59037413046145 topic_num = 3, coherence = -10.51994121341506, perplexity = 8.421586281561325 topic_num = 4, coherence = -10.498935784230891, perplexity = 9.163865911812838 topic_num = 5, coherence = -10.466505553089613, perplexity = 10.02873590975954 topic_num = 6, coherence = -10.427246025202495, perplexity = 10.706792157460887 topic_num = 7, coherence = -10.441670962642908, perplexity = 11.007513545383127 topic_num = 8, coherence = -10.431903836350067, perplexity = 11.393319548027026 topic_num = 9, coherence = -10.394974053783624, perplexity = 13.154796351781842 topic_num = 10, coherence = -10.398193229861565, perplexity = 13.453254319022557 topic_num = 11, coherence = -10.393056192535115, perplexity = 13.771475137747052 topic_num = 12, coherence = -10.386759634629335, perplexity = 14.178980599173155 topic_num = 13, coherence = -10.395241748738718, perplexity = 16.132824693572804 ・・・ num_topics = 18, coherence = -10.373039938078676, perplexity = 21.76790238689796 num_topics = 19, coherence = -10.336482759968458, perplexity = 20.067649661316306 topic_num = 20, coherence = -10.318518756029693, perplexity = 21.38207737535069 ・・・ num_topics = 28, coherence = -10.297976846891006, perplexity = 25.2833756062596 num_topics = 29, coherence = -10.279231366719717, perplexity = 26.40726049105775 num_topics = 30, coherence = -10.266658546693755, perplexity = 26.52593230907953
(3) 結果の出力
show_topics
show_topics もしくは print_topics でトピックの内容をログ出力します。
ログ出力を有効化していなくても、ログへの出力内容をメソッドの戻り値として取得できます。
topic_num = 8 alpha = 0.01 lda = LdaModel(corpus = corpus, id2word = dic, num_topics = topic_num, alpha = alpha, random_state = 1) for t in lda.show_topics(): print(t)
出力結果
(0, '0.259*"C" + 0.131*"T" + 0.100*"N" + 0.068*"O" + 0.068*"F" + 0.068*"P" + 0.068*"B" + 0.036*"D" + 0.036*"W" + 0.036*"R"') (1, '0.234*"B" + 0.159*"D" + 0.084*"O" + 0.084*"W" + 0.084*"G" + 0.084*"C" + 0.084*"R" + 0.084*"S" + 0.009*"H" + 0.009*"F"') (2, '0.223*"C" + 0.149*"S" + 0.094*"R" + 0.076*"B" + 0.076*"F" + 0.076*"P" + 0.076*"D" + 0.057*"N" + 0.057*"W" + 0.039*"L"') (3, '0.172*"S" + 0.172*"N" + 0.172*"C" + 0.172*"R" + 0.091*"Y" + 0.091*"B" + 0.010*"F" + 0.010*"G" + 0.010*"A" + 0.010*"H"') (4, '0.223*"B" + 0.113*"R" + 0.094*"S" + 0.094*"P" + 0.076*"W" + 0.076*"D" + 0.076*"Y" + 0.057*"C" + 0.057*"M" + 0.039*"T"') (5, '0.191*"S" + 0.173*"B" + 0.139*"M" + 0.105*"C" + 0.088*"F" + 0.088*"W" + 0.071*"N" + 0.036*"D" + 0.036*"R" + 0.019*"T"') (6, '0.195*"O" + 0.163*"S" + 0.100*"R" + 0.100*"W" + 0.068*"P" + 0.068*"L" + 0.068*"B" + 0.036*"Y" + 0.036*"U" + 0.036*"T"') (7, '0.241*"W" + 0.163*"O" + 0.163*"B" + 0.084*"H" + 0.084*"C" + 0.044*"T" + 0.044*"N" + 0.044*"G" + 0.044*"R" + 0.044*"U"')
get_topic_terms
上記は加工された文字列でしたが、get_topic_terms でトピック内の単語とその確率を取得できます。
topn (デフォルトは 10) でトピック内の単語を確率の高い順にいくつ取得するかを指定できます。
from toolz import frequencies # 文書毎の該当トピック doc_topics = [lda[c] for c in corpus] # トピックの該当数 topic_freq = frequencies([t[0] for dt in doc_topics for t in dt]) for i in range(topic_num): items = [(dic[t[0]], t[1]) for t in lda.get_topic_terms(i, topn = 5)] print(f"topic_id = {i}, freq = {topic_freq[i]}, items = {items}")
出力結果
topic_id = 0, freq = 10, items = [('C', 0.25896409), ('T', 0.13147409), ('N', 0.099601582), ('P', 0.067729078), ('F', 0.067729078)] topic_id = 1, freq = 4, items = [('B', 0.23364486), ('D', 0.15887851), ('O', 0.084112152), ('W', 0.084112152), ('G', 0.084112152)] topic_id = 2, freq = 11, items = [('C', 0.22298852), ('S', 0.14942528), ('R', 0.094252877), ('P', 0.075862072), ('B', 0.075862072)] topic_id = 3, freq = 5, items = [('S', 0.17171718), ('N', 0.17171718), ('C', 0.17171715), ('R', 0.17171715), ('Y', 0.090909086)] topic_id = 4, freq = 14, items = [('B', 0.22298847), ('R', 0.11264367), ('S', 0.094252862), ('P', 0.094252862), ('W', 0.075862065)] topic_id = 5, freq = 17, items = [('S', 0.19057818), ('B', 0.17344755), ('M', 0.13918629), ('C', 0.10492505), ('F', 0.087794438)] topic_id = 6, freq = 8, items = [('O', 0.1952191), ('S', 0.16334662), ('W', 0.099601582), ('R', 0.099601582), ('P', 0.067729086)] topic_id = 7, freq = 8, items = [('W', 0.24137934), ('O', 0.1625616), ('B', 0.1625616), ('H', 0.083743848), ('C', 0.083743848)]
get_document_topics
上記ではトピックの構成を取得しましたが、get_document_topics で per_word_topics = True を指定すると、文書内の単語がどのトピックへどの程度の確率で該当するかを取得できます。
for i in range(len(corpus)): dts = lda.get_document_topics(corpus[i], per_word_topics = True) for dt in dts[2]: item = dic[dt[0]] print(f"corpus = {i}, item = {item}, topic_id = {dt[1]}")
出力結果
corpus = 0, item = C, topic_id = [(5, 1.0000001)] corpus = 0, item = M, topic_id = [(5, 1.0)] corpus = 0, item = R, topic_id = [(5, 1.0000001)] corpus = 0, item = S, topic_id = [(5, 0.99999994)] corpus = 1, item = C, topic_id = [(0, 1.0000001)] corpus = 1, item = T, topic_id = [(0, 1.0)] corpus = 1, item = Y, topic_id = [(0, 1.0)] corpus = 2, item = C, topic_id = [(4, 1.0)] corpus = 2, item = M, topic_id = [(4, 1.0)] corpus = 2, item = Y, topic_id = [(4, 1.0)] corpus = 2, item = P, topic_id = [(4, 1.0000001)] corpus = 3, item = L, topic_id = [(6, 1.0)] corpus = 3, item = O, topic_id = [(6, 1.0)] corpus = 3, item = W, topic_id = [(6, 0.99999994)] ・・・ corpus = 7, item = C, topic_id = [(0, 0.77635038), (4, 0.22364961)] corpus = 7, item = T, topic_id = [(0, 0.72590601), (4, 0.27409402)] corpus = 7, item = Y, topic_id = [(0, 0.1830055), (4, 0.81699443)] corpus = 7, item = W, topic_id = [(0, 0.1830055), (4, 0.81699443)] corpus = 7, item = B, topic_id = [(0, 0.14557858), (4, 0.85442138)] corpus = 7, item = D, topic_id = [(0, 0.1830055), (4, 0.81699443)] corpus = 7, item = F, topic_id = [(0, 0.74502456), (4, 0.25497544)] ・・・
(4) 可視化
pyLDAvis を使うと LdaModel のトピック内容を可視化できます。
pyLDAvis を使うには予め pip 等でインストールしておきます。
インストール例
> pip install pyldavis
可視化1(PCoA)
以下のような処理で Jupyter Notebook 上に D3.js で可視化した結果を表示できます。
トピックの番号は 1 から始まり、デフォルトではソートされてしまう点に注意。
トピックをソートさせたくない(LdaModel 内と同じ順序にしたい)場合は sort_topics = False を指定します。
import pyLDAvis.gensim vis = pyLDAvis.gensim.prepare(lda, corpus, dic, n_jobs = 1, sort_topics = False) pyLDAvis.display(vis)
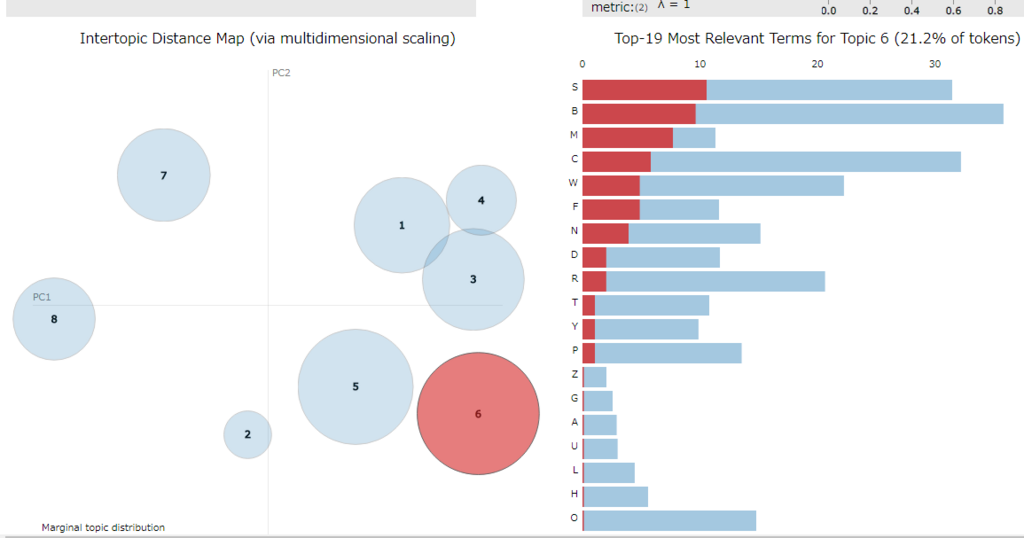
可視化2(Metric MDS)
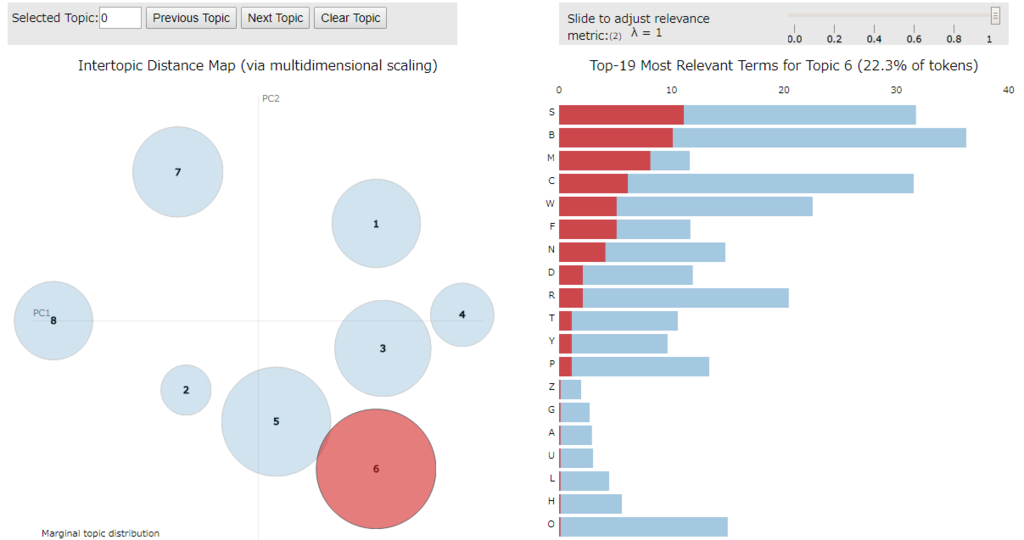
次元削減の方法としてデフォルトでは PCoA(主座標分析)を使うようですが、mds パラメータで変更できます。
以下は mmds を指定し Metric MDS(多次元尺度構成法) にしてみました。
import pyLDAvis.gensim vis = pyLDAvis.gensim.prepare(lda, corpus, dic, n_jobs = 1, mds='mmds', sort_topics = False) pyLDAvis.display(vis)
HTML ファイル化
pyLDAvis の結果を HTML ファイルへ保存したい場合は以下のようにします。
import pyLDAvis.gensim vis = pyLDAvis.gensim.prepare(lda, corpus, dic, n_jobs = 1, sort_topics = False) pyLDAvis.save_html(vis, 'sample.html')
3. LDA の結果
(a) 併売の組み合わせとトピックの内容
出現数の多い順に(2つの)組み合わせが該当するトピックを抽出してみます。
from itertools import combinations from toolz import unique cmb = frequencies([c for s in sentences for c in combinations(sorted(unique(s)), 2)]) for (k1, k2), v in sorted(cmb.items(), key = lambda x: -x[1]): topics = lda[dic.doc2bow([k1, k2])] print(f"item1 = {k1}, item2 = {k2}, freq = {v}, topics = {topics}")
結果は以下の通りです。
B と S の組み合わせは トピック 5、C と S の組み合わせは トピック 2 で最も確率の高い組み合わせになっていますし、その他も概ねトピック内の確率が高めになっているように見えます。
実行結果
item1 = B, item2 = S, freq = 16, topics = [(5, 0.9663462)] item1 = C, item2 = S, freq = 14, topics = [(2, 0.9663462)] item1 = R, item2 = S, freq = 13, topics = [(3, 0.9663462)] item1 = B, item2 = C, freq = 12, topics = [(5, 0.9663462)] item1 = B, item2 = W, freq = 12, topics = [(7, 0.9663462)] item1 = C, item2 = R, freq = 10, topics = [(2, 0.9663462)] item1 = O, item2 = W, freq = 10, topics = [(7, 0.9663462)] item1 = C, item2 = N, freq = 10, topics = [(0, 0.9663462)] item1 = S, item2 = W, freq = 10, topics = [(5, 0.9663462)] item1 = C, item2 = W, freq = 9, topics = [(7, 0.9663462)] item1 = B, item2 = R, freq = 9, topics = [(4, 0.9663462)] item1 = C, item2 = T, freq = 8, topics = [(0, 0.9663462)] item1 = C, item2 = D, freq = 8, topics = [(2, 0.9663462)] item1 = C, item2 = F, freq = 8, topics = [(0, 0.9663462)] item1 = R, item2 = W, freq = 8, topics = [(4, 0.9663462)] item1 = O, item2 = R, freq = 7, topics = [(6, 0.9663462)] item1 = B, item2 = D, freq = 7, topics = [(1, 0.9663462)] item1 = B, item2 = P, freq = 7, topics = [(4, 0.9663462)] item1 = B, item2 = M, freq = 7, topics = [(5, 0.9663462)] item1 = C, item2 = Y, freq = 6, topics = [(3, 0.9663462)] item1 = C, item2 = P, freq = 6, topics = [(0, 0.9663462)] item1 = B, item2 = O, freq = 6, topics = [(7, 0.9663462)] item1 = B, item2 = F, freq = 6, topics = [(5, 0.9663462)] item1 = B, item2 = Y, freq = 6, topics = [(4, 0.9663462)] item1 = D, item2 = W, freq = 6, topics = [(1, 0.9663462)] item1 = B, item2 = N, freq = 6, topics = [(5, 0.9663462)] item1 = N, item2 = S, freq = 6, topics = [(3, 0.9663462)] item1 = P, item2 = S, freq = 6, topics = [(2, 0.9663462)] item1 = D, item2 = S, freq = 6, topics = [(1, 0.9663462)] ・・・
(b) アソシエーション分析の結果とトピックの内容
アソシエーション分析で抽出した組み合わせに対するトピックも抽出してみます。
for c in [['B', 'O', 'W'], ['B', 'T', 'C'], ['N', 'C'], ['T', 'C']]: topics = lda[dic.doc2bow(c)] print(f"items = {c}, topics = {topics}")
こちらもトピックへ概ね反映できているように見えます。
実行結果
items = ['B', 'O', 'W'], topics = [(7, 0.97727275)] items = ['B', 'T', 'C'], topics = [(0, 0.97727275)] items = ['N', 'C'], topics = [(3, 0.9663462)] items = ['T', 'C'], topics = [(0, 0.9663462)]
(c) 未知の組み合わせ
トピックモデルの場合、データセットに無いが潜在的に確率の高そうな組み合わせを抽出する事も可能だと思われます。(有用かどうかは分かりませんが)
例えば、以下のような処理でデータセットに無い組み合わせで確率の高いものから順に 5件抽出してみました。
from toolz import topk # データセットの組み合わせ ds = [c for s in sentences for c in combinations(sorted(s), 2)] topic_terms = lambda i: [(dic[t[0]], t[1]) for t in lda.get_topic_terms(i)] # トピック毎のデータセットに無い組み合わせ ts = [ ((t1[0], t2[0]), t1[1] * t2[1]) for i in range(topic_num) for t1, t2 in combinations(sorted(topic_terms(i), key = lambda x: x[0]), 2) if (t1[0], t2[0]) not in ds ] for (k1, k2), v in topk(5, ts, key = lambda x: x[1]): print(f"items1 = {k1}, items2 = {k2}, score = {v}")
実行結果
items1 = C, items2 = G, score = 0.007074854336678982 items1 = G, items2 = R, score = 0.007074854336678982 items1 = G, items2 = S, score = 0.007074854336678982 items1 = O, items2 = Y, score = 0.0069998884573578835 items1 = S, items2 = U, score = 0.00585705041885376
IDWR データで再帰型ニューラルネットワーク - Keras
前回 加工した IDWR データ を使って再帰型ニューラルネットワーク(RNN)を Keras + TensorFlow で試してみました。
ソースは http://github.com/fits/try_samples/tree/master/blog/20180121/
はじめに
インストール
tensorflow と keras をインストールしておきます。
インストール例
> pip install tensorflow > pip install keras
データセット
データセットは 前回 に作成した、2014年 1週目 ~ 2017年 49週目 (2015年は 53週目まで) の teiten.csv を 1つの csv ファイル (idwr.csv) へ連結したものを使います。
再帰型ニューラルネットワーク(RNN)
指定の感染症に対する週別の全国合計値を再帰型ニューラルネットワーク(RNN)で学習・予測する処理を実装してみます。
(a) 4週分のデータを使用
とりあえず、任意の週の過去 4週の報告数を入力データ、その週の報告数をラベルデータとして学習を実施してみます。
ただ、入力データに季節性(周期性)の指標となるデータ(ここでは週)を含んでいないので、季節性のあるデータに対する結果は期待できないように思います。
データ加工
入力データとラベルデータは、以下の要領で 1週分ずつスライドしたものを用意します。
- 2014年 1 ~ 4週目を入力データとし 5週目をラベルデータ
- 2014年 2 ~ 5週目を入力データとし 6週目をラベルデータ
入力データ・ラベルデータの作成例(インフルエンザ)
import pandas as pd import numpy as np t = 4 df = pd.read_csv('idwr.csv', encoding = 'UTF-8') ds = df.groupby(['year', 'week'])['インフルエンザ'].sum() def window(d, wsize): return [d[i:(i + wsize)].values.flatten() for i in range(len(d) - wsize + 1)] # t + 1 週分のスライドデータを作成 dw = window(ds.astype('float'), t + 1) # 入力データ(t週分) data = np.array([i[0:-1] for i in dw]).reshape(len(dw), t, 1) # ラベルデータ(残り 1週分) labels = np.array([i[-1] for i in dw]).reshape(len(dw), 1)
上記では 1週分ずつスライドさせた 5週分の配列を作成し、4週分を入力データ、残りの 1週分をラベルデータとしています。
その結果、data・labels 変数の内容は以下のようになります。
data 変数の内容
array([[[ 9.89100000e+03], [ 2.71000000e+04], [ 5.82330000e+04], [ 1.22618000e+05]], [[ 2.71000000e+04], [ 5.82330000e+04], [ 1.22618000e+05], [ 1.70403000e+05]], [[ 5.82330000e+04], [ 1.22618000e+05], [ 1.70403000e+05], [ 1.51829000e+05]], ・・・ [[ 2.40700000e+03], [ 2.58800000e+03], [ 3.79900000e+03], [ 7.28000000e+03]], [[ 2.58800000e+03], [ 3.79900000e+03], [ 7.28000000e+03], [ 1.27850000e+04]]])
labels 変数の内容
array([[ 1.70403000e+05], [ 1.51829000e+05], [ 1.39162000e+05], ・・・ [ 1.27850000e+04], [ 2.01270000e+04]])
学習
上記のデータを使って再帰型ニューラルネットワークの学習処理を実施してみます。
今回は GRU(ゲート付き再帰的ユニット)を使いました。
学習処理例(Jupyter Notebook 使用)
%matplotlib inline import matplotlib.pyplot as plt from keras.models import Sequential from keras.layers.core import Dense, Activation from keras.layers.recurrent import GRU from keras.optimizers import Nadam epoch = 3000 batch_size = 52 n_num = 80 model = Sequential() # 再帰型ニューラルネットワークの GRU 使用 model.add(GRU(n_num, activation = 'relu', input_shape = (t, 1))) model.add(Dense(1)) model.add(Activation('linear')) opt = Nadam() # モデルの構築 model.compile(loss = 'mean_squared_error', optimizer = opt) # 学習 hist = model.fit(data, labels, epochs = epoch, batch_size = batch_size) # 誤差の遷移状況をグラフ化 plt.plot(hist.history['loss'])
処理結果は以下の通り。 誤差の遷移を見る限り、学習は進んでいるように見えます。
学習処理結果例
Epoch 1/3000 202/202 [==============================] - 1s 7ms/step - loss: 3604321859.1683 Epoch 2/3000 202/202 [==============================] - 0s 193us/step - loss: 3120768191.3663 ・・・ Epoch 2999/3000 202/202 [==============================] - 0s 183us/step - loss: 41551096.5149 Epoch 3000/3000 202/202 [==============================] - 0s 193us/step - loss: 41367050.6931

予測
3つのグラフを描画して学習したモデルの予測能力を比べてみます。
| 名称 | 概要 |
|---|---|
| actual | 実データ |
| predict1 | 実データを入力データにした予測結果(学習時の入力データをそのまま使用) |
| predict2 | 実データの先頭のみを使った予測結果 |
predict2 は以下のように最初だけ実データを使って、予測結果を次の入力データの一部にして予測を繋げていった結果です。
- 2014年 1 ~ 4週目を入力データとし 5週目を予測
- 2014年 2 ~ 4週目と 5週目の予測値を入力データとし 6週目を予測
- 2014年 3 ~ 4週目と 5 ~ 6週目の予測値を入力データとし 7週目を予測
- 2014年 4週目と 5 ~ 7週目の予測値を入力データとし 8週目を予測
- 5 ~ 8週目の予測値を入力データとし 9週目を予測
そのため、実際の予測では predict2 の結果が重要となります。
予測処理例(Jupyter Notebook 使用)
from functools import reduce # 実データの描画 plt.plot(ds.values, label = 'actual') # 入力データを使った予測(predict1) res1 = model.predict(data) # predict1 の結果を描画 plt.plot(range(t, len(res1) + t), res1, label = 'predict1') # predict2 用の予測処理 def predict(a, b): r = model.predict(a[1]) return ( np.append(a[0], r), np.append(a[1][:, 1:], np.array([r]), axis = 1) ) # 入力データの先頭 fst_data = data[0].reshape(1, t, 1) # 入力データの先頭を使った予測(predict2) res2, _ = reduce(predict, range(len(ds) - t), (np.array([]), fst_data)) # predict2 の結果を描画 plt.plot(range(t, len(res2) + t), res2, label = 'predict2') plt.legend()
インフルエンザのデータに対して適用した結果が以下です。
予測結果(インフルエンザ)

predict1 は実データをほぼ再現できていますが、predict2 は全く駄目でした。
やはり、入力データへ週の情報を与えなかった事が原因だと思われます。
備考
sample1.py
import sys import matplotlib.pyplot as plt import numpy as np import pandas as pd from functools import reduce from keras.models import Sequential from keras.layers.core import Dense, Activation from keras.layers.recurrent import GRU from keras.optimizers import Nadam data_file = sys.argv[1] item_name = sys.argv[2] dest_file = sys.argv[3] t = 4 epoch = 3000 batch_size = 52 n_num = 80 df = pd.read_csv(data_file, encoding = 'UTF-8') ds = df.groupby(['year', 'week'])[item_name].sum() def window(d, wsize): return [d[i:(i + wsize)].values.flatten() for i in range(len(d) - wsize + 1)] dw = window(ds.astype('float'), t + 1) data = np.array([i[0:-1] for i in dw]).reshape(len(dw), t, 1) labels = np.array([i[-1] for i in dw]).reshape(len(dw), 1) model = Sequential() model.add(GRU(n_num, activation = 'relu', input_shape = (t, 1))) model.add(Dense(1)) model.add(Activation('linear')) opt = Nadam() model.compile(loss = 'mean_squared_error', optimizer = opt) hist = model.fit(data, labels, epochs = epoch, batch_size = batch_size) fig, axes = plt.subplots(1, 2, figsize = (12, 6)) axes[0].plot(hist.history['loss']) axes[1].plot(ds.values, label = 'actual') res1 = model.predict(data) axes[1].plot(range(t, len(res1) + t), res1, label = 'predict1') def predict(a, b): r = model.predict(a[1]) return ( np.append(a[0], r), np.append(a[1][:, 1:], np.array([r]), axis = 1) ) fst_data = data[0].reshape(1, t, 1) res2, _ = reduce(predict, range(len(ds) - t), (np.array([]), fst_data)) axes[1].plot(range(t, len(res2) + t), res2, label = 'predict2') axes[1].legend() plt.savefig(dest_file)
(b) 年と週を追加したデータを使用
季節性の強いインフルエンザのようなデータに対して (a) の入力データでは無理があった気がするので、次は入力データへ年と週を追加して試してみます。
入力データとラベルデータ
(a) の入力データへラベルデータの年と週を追加します。
- 2014 と 5、2014年 1 ~ 4週目を入力データとし 5週目をラベルデータ
- 2014 と 6、2014年 2 ~ 5週目を入力データとし 6週目をラベルデータ
入力データ(data 変数の内容)例
array([[[ 2.01400000e+03], [ 5.00000000e+00], [ 9.89100000e+03], [ 2.71000000e+04], [ 5.82330000e+04], [ 1.22618000e+05]], [[ 2.01400000e+03], [ 6.00000000e+00], [ 2.71000000e+04], [ 5.82330000e+04], [ 1.22618000e+05], [ 1.70403000e+05]], ・・・ [[ 2.01700000e+03], [ 4.80000000e+01], [ 2.40700000e+03], [ 2.58800000e+03], [ 3.79900000e+03], [ 7.28000000e+03]], [[ 2.01700000e+03], [ 4.90000000e+01], [ 2.58800000e+03], [ 3.79900000e+03], [ 7.28000000e+03], [ 1.27850000e+04]]])
ラベルデータ(labels 変数の内容)例
array([[ 1.70403000e+05], [ 1.51829000e+05], ・・・ [ 1.27850000e+04], [ 2.01270000e+04]])
学習と予測処理
発症報告数と週の値では桁数が大きく異なるケースがあるので、このままで効果的な学習ができるかは微妙です。
そこで、ここではバッチ正規化(BatchNormalization)の層を先頭に追加する事で対処してみました。
バッチ正規化によりミニバッチ単位でデータの正規化が行われるようになります。 この場合、ミニバッチのサイズが重要になってくると思います。
また、IDWR では ISO 週番号を使っているようなので、年によって 52週の場合と 53週の場合があります。
ここでは isocalendar でその年の最終週を取得する事で対応しました。(12/28 を含む週がその年の最終週)
sample2.py
import sys import matplotlib.pyplot as plt import numpy as np import pandas as pd from datetime import datetime from functools import reduce from keras.models import Sequential from keras.layers.core import Dense, Activation from keras.layers.normalization import BatchNormalization from keras.layers.recurrent import GRU from keras.optimizers import Nadam data_file = sys.argv[1] item_name = sys.argv[2] dest_file = sys.argv[3] predict_size = int(sys.argv[4]) batch_size = int(sys.argv[5]) t = 4 epoch = 5000 n_num = 80 df = pd.read_csv(data_file, encoding = 'UTF-8') ds = df.groupby(['year', 'week'])[item_name].sum() def window_with_index(d, wsize): return [ np.r_[ d.index[i + wsize - 1][0], # 年 d.index[i + wsize - 1][1], # 週 d[i:(i + wsize)].values.flatten() ] for i in range(len(d) - wsize + 1) ] dw = window_with_index(ds.astype('float'), t + 1) # 入力データの要素数 input_num = len(dw[0]) - 1 data = np.array([i[0:-1] for i in dw]).reshape(len(dw), input_num, 1) labels = np.array([i[-1] for i in dw]).reshape(len(dw), 1) model = Sequential() # バッチ正規化 model.add(BatchNormalization(axis = 1, input_shape = (input_num, 1))) model.add(GRU(n_num, activation = 'relu')) model.add(Dense(1)) model.add(Activation('linear')) opt = Nadam() model.compile(loss = 'mean_squared_error', optimizer = opt) hist = model.fit(data, labels, epochs = epoch, batch_size = batch_size) fig, axes = plt.subplots(1, 2, figsize = (12, 6)) axes[0].plot(hist.history['loss']) # 実データの描画 axes[1].plot(ds.values, label = 'actual') # predict1(入力データをそのまま使用) res1 = model.predict(data) axes[1].plot(range(t, len(res1) + t), res1, label = 'predict1') # 指定年の最終週(52 か 53)の取得 def weeks_of_year(year): return datetime(year, 12, 28).isocalendar()[1] # predict2 用の予測処理 def predict(a, b): r = model.predict(a[1]) year = a[1][0, 0, 0] week = a[1][0, 1, 0] + 1 if week > weeks_of_year(int(year)): year += 1 week = 1 next = np.r_[ year, week, a[1][:, 3:].flatten(), r.flatten() ].reshape(a[1].shape) return (np.append(a[0], r), next) fst_data = data[0].reshape(1, input_num, 1) # predict2(入力データの先頭のみ使用) res2, _ = reduce(predict, range(predict_size), (np.array([]), fst_data)) axes[1].plot(range(t, predict_size + t), res2, label = 'predict2') min_year = min(ds.index.levels[0]) years = range(min_year, min_year + int(predict_size / 52) + 1) # X軸のラベル設定 axes[1].set_xticklabels(years) # X軸の目盛設定 axes[1].set_xticks( reduce(lambda a, b: a + [a[-1] + weeks_of_year(b)], years[:-1], [0]) ) # 凡例をグラフ外(下部)へ横並びで出力 axes[1].legend(bbox_to_anchor = (1, -0.1), ncol = 3) fig.subplots_adjust(bottom = 0.15) plt.savefig(dest_file)
実行
インフルエンザに対して 2014年 5週目から 250週分をバッチサイズ 52 で予測(predict2)するように実行してみます。
実行例(インフルエンザ)
> python sample2.py idwr.csv インフルエンザ sample2.png 250 52 ・・・ Epoch 4998/5000 202/202 [==============================] - 0s 155us/step - loss: 133048118.4158 Epoch 4999/5000 202/202 [==============================] - 0s 155us/step - loss: 154247298.2970 Epoch 5000/5000 202/202 [==============================] - 0s 232us/step - loss: 171130778.4554
1. インフルエンザ (250週予測, batch_size = 52)

predict2 は劇的に改善され、将来の予測値もそれっぽいものになりました。
他の感染症に対しても同じように処理してみると以下のようになりました。
2. 感染性胃腸炎 (250週予測, batch_size = 52)

3. 手足口病1 (250週予測, batch_size = 52)

2年周期を学習できていないような結果となりました。
そこで、バッチサイズを 104 にして 320週分を予測してみたところ以下のようになりました。
4. 手足口病2 (320週予測, batch_size = 104)

5. 水痘1 (250週予測, batch_size = 52)

predict2 の差異が目立っています。
そこで、バッチサイズを 26 にしてみたところ改善が見られました。 ちなみに、バッチサイズを大きくしても特に改善は見られませんでした。
6. 水痘2 (250週予測, batch_size = 26)

7. 流行性耳下腺炎 (250週予測, batch_size = 52)

IDWR データの入手と加工
時系列データ分析を試すのに適した季節性(周期性)を持つオープンデータを探していて以下を見つけました。
インフルエンザ等の感染症の週単位の報告数が都道府県別にまとまっており、csv ファイルでデータを入手できるようになっています。
今回は、上記データを分析するための前処理(入手と加工)を行います。
ソースは http://github.com/fits/try_samples/tree/master/blog/20180114/
(1) データ入手
「定点把握疾患(週報告)、報告数、定点当たり報告数、都道府県別」の場合、csv ファイル名は <年4桁>-<週2桁>-teiten.csv となっています。
週は 1 ~ 52 もしくは 53 となっており、年によって 52 の場合と 53 の場合があります。
URL のルールが決まっているので過去データも簡単にダウンロードできましたが、curl コマンド等を使ってダウンロードする際は HTTP リクエストヘッダーへ User-Agent を付ける必要がありました。
teiten.csv 内容
teiten.csv の内容は以下の通りで、文字コードは Shift_JIS となっています。
これをそのままデータ分析で使うには以下の点が気になります。
- ヘッダー行が複数行
- 報告がない場合は
-となっている - 最終行が空データ
2017-44-teiten.csv
"報告数・定点当り報告数、疾病・都道府県別","","","","","","","","","","","","","","","","","","","","","","","","","","","","","","","","","","","","","","" "2017年44週(10月30日~11月05日)","2017年11月08日作成","","","","","","","","","","","","","","","","","","","","","","","","","","","","","","","","","","","","","" "","インフルエンザ","","RSウイルス感染症","","咽頭結膜熱","","A群溶血性レンサ球菌咽頭炎","","感染性胃腸炎","","水痘","","手足口病","","伝染性紅斑","","突発性発しん","","百日咳","","ヘルパンギーナ","","流行性耳下腺炎","","急性出血性結膜炎","","流行性角結膜炎","","細菌性髄膜炎","","無菌性髄膜炎","","マイコプラズマ肺炎","","クラミジア肺炎","","感染性胃腸炎(ロタウイルス)","" "","報告","定当","報告","定当","報告","定当","報告","定当","報告","定当","報告","定当","報告","定当","報告","定当","報告","定当","報告","定当","報告","定当","報告","定当","報告","定当","報告","定当","報告","定当","報告","定当","報告","定当","報告","定当","報告","定当" "総数","2407","0.49","3033","","1621","0.51","5940","1.88","10937","3.47","1469","0.47","5126","1.62","173","0.05","1259","0.40","50","0.02","967","0.31","899","0.28","8","0.01","484","0.70","9","0.02","11","0.02","185","0.39","5","0.01","4","0.01" "北海道","137","0.62","120","","369","2.65","384","2.76","217","1.56","71","0.51","103","0.74","2","0.01","33","0.24","5","0.04","9","0.06","11","0.08","-","-","9","0.31","-","-","1","0.04","9","0.39","-","-","1","0.04" "青森県","10","0.15","35","","12","0.29","44","1.05","74","1.76","21","0.50","121","2.88","-","-","11","0.26","1","0.02","11","0.26","28","0.67","-","-","4","0.36","-","-","-","-","5","0.83","-","-","-","-" ・・・ "鹿児島県","79","0.87","37","","46","0.87","76","1.43","281","5.30","20","0.38","134","2.53","-","-","20","0.38","-","-","20","0.38","81","1.53","-","-","6","0.86","-","-","-","-","2","0.17","-","-","-","-" "沖縄県","230","3.97","13","","16","0.47","25","0.74","71","2.09","7","0.21","42","1.24","-","-","10","0.29","3","0.09","7","0.21","3","0.09","-","-","13","1.44","-","-","-","-","-","-","1","0.14","-","-" "","","","","","","","","","","","","","","","","","","","","","","","","","","","","","","","","","","","","","",""

(2) 加工
複数の teiten.csv から必要な情報のみを抽出して単一の csv へ変換する処理を Python で実装してみました。
基本的な処理は pandas.read_csv で行っています。
| pandas.read_csv の引数 | 内容 |
|---|---|
| skiprows | 不要なヘッダー行と総数の行を除外(総数は算出できるため) |
| skipfooter | 最終行の空データを除外 |
| usecols | "定当" 列を除外 |
| converters | '-' を 0 へ変換 |
時系列データ分析用に年と週、そして週の最終日を追加しています。
idwr_convert.py
# coding: utf-8 import codecs import functools import glob import os import re import sys import pandas as pd data_files = f"{sys.argv[1]}/*.csv" dest_file = sys.argv[2] r = re.compile('"([0-9]+)年([0-9]+)週\(.*[^0-9]([0-9]+)月([0-9]+)日\)') # 抽出する列(定当データの除外) cols = [i for i in range(38) if i == 0 or i % 2 == 1] # 変換処理('-' を 0 へ変換) conv = lambda x: 0 if x == '-' else int(x) # 列毎の変換処理 conv_map = {i:conv for i in range(len(cols)) if i > 0} # 年、週、その週の最終日を抽出 def read_info(file): f = codecs.open(file, 'r', 'Shift_JIS') f.readline() m = r.match(f.readline()) f.close() year = int(m.group(1)) week = int(m.group(2)) # 50週を超えていて 1月なら次の年 last_year = year + 1 if week > 50 and m.group(3) == '01' else year return (year, week, f"{last_year}-{m.group(3)}-{m.group(4)}") def read_csv(file): d = pd.read_csv(file, encoding = 'Shift_JIS', skiprows = [0, 1, 3, 4], usecols = cols, skipfooter = 1, converters = conv_map, engine = 'python') info = read_info(file) d['year'] = info[0] # 年 d['week'] = info[1] # 週 d['lastdate'] = info[2] # その週の最終日 return d.rename(columns = {'Unnamed: 0': 'pref'}) dfs = [read_csv(f) for f in glob.glob(data_files)] # データフレームの連結 df = functools.reduce(lambda a, b: a.append(b), dfs) # csv ファイルとして出力 df.to_csv(dest_file, encoding = 'UTF-8')
実行
2014年 1週目 ~ 2017年 49週目 (2015年は 53週目まである) の teiten.csv を data ディレクトリを配置して、上記を実行しました。
実行例
> python idwr_convert.py data idwr.csv
idwr.csv 例
,pref,インフルエンザ,RSウイルス感染症,咽頭結膜熱,A群溶血性レンサ球菌咽頭炎,感染性胃腸炎,水痘,手足口病,伝染性紅斑,突発性発しん,百日咳,ヘルパンギーナ,流行性耳下腺炎,急性出血性結膜炎,流行性角結膜炎,細菌性髄膜炎,無菌性髄膜炎,マイコプラズマ肺炎,クラミジア肺炎,感染性胃腸炎(ロタウイルス),year,week,lastdate 0,北海道,333,125,40,118,203,236,2,2,12,0,5,4,0,1,0,0,0,0,1,2014,1,2014-01-05 1,青森県,77,22,18,9,134,71,0,10,4,0,0,7,0,0,0,0,0,0,2,2014,1,2014-01-05 2,岩手県,98,17,11,25,224,82,2,3,7,0,0,15,0,7,0,1,10,0,1,2014,1,2014-01-05 ・・・ 44,宮崎県,20,47,13,58,191,23,9,18,15,0,1,55,0,11,0,0,0,0,0,2015,53,2016-01-03 45,鹿児島県,40,27,50,109,303,40,4,32,21,0,0,63,0,7,0,0,0,0,0,2015,53,2016-01-03 46,沖縄県,353,3,5,30,209,30,1,2,7,3,1,57,0,2,1,1,9,0,0,2015,53,2016-01-03 0,北海道,1093,147,116,491,433,101,17,273,39,2,1,306,0,6,1,0,11,0,11,2016,1,2016-01-10 1,青森県,142,31,23,52,201,14,0,24,14,0,0,38,0,7,2,1,3,0,0,2016,1,2016-01-10 2,岩手県,156,33,4,125,255,14,2,15,18,1,2,29,0,13,1,0,14,0,0,2016,1,2016-01-10 ・・・ 44,宮崎県,347,46,81,98,358,14,40,0,29,2,4,24,0,15,0,1,0,0,0,2017,49,2017-12-10 45,鹿児島県,252,24,83,134,426,38,71,1,18,0,11,86,0,8,0,0,0,0,0,2017,49,2017-12-10 46,沖縄県,410,5,8,44,70,24,72,2,9,0,6,4,0,20,0,1,0,0,0,2017,49,2017-12-10
(3) グラフ化
idwr.csv を感染症毎に集計して折れ線グラフ化してみます。
(a) matplotlib 使用 - 全体
まずは matplotlib を使って全ての感染症を同一グラフへ表示してみます。
idwr_plot_matplotlib.py
import sys import pandas as pd import matplotlib.pyplot as plt data_file = sys.argv[1] img_file = sys.argv[2] df = pd.read_csv(data_file, parse_dates = ['lastdate']) df.groupby('lastdate').sum().iloc[:, 1:20].plot(legend = False) plt.savefig(img_file)
ここで、df.groupby('lastdate').sum() では lastdate 毎にグルーピングして合計しています。

これだと不要な列も含んでしまうので df.groupby('lastdate').sum().iloc[:, 1:20] で必要な列のみを抽出します。

実行結果は以下の通りです。
実行例
> python idwr_plot_matplotlib.py idwr.csv idwr_matplotlib.png
idwr_matplotlib.png 例

他と比べ目立って報告数が多いのはインフルエンザです。
(b) matplotlib 使用 - 個別
次に指定したものだけをグラフ表示してみます。
idwr_plot_matplotlib2.py
import sys import pandas as pd import matplotlib.pyplot as plt data_file = sys.argv[1] item_name = sys.argv[2] img_file = sys.argv[3] df = pd.read_csv(data_file, parse_dates = ['lastdate']) df.groupby('lastdate').sum()[item_name].plot(legend = False) plt.savefig(img_file)
実行例
> python idwr_plot_matplotlib2.py idwr.csv インフルエンザ インフルエンザ.png
グラフ例
グラフ形状的に特徴のあるものをいくつかピックアップしてみました。
1. インフルエンザ
1年周期で一定期間内に大流行しています。

2. 感染性胃腸炎
1年周期ですが、2016年の末頃は(異常に)大流行しています。

3. 手足口病
2年周期で流行しています。

4. 水痘
なんとなく 1年周期がありそうですが、全体的な傾向は下がっているように見えます。

5. 流行性耳下腺炎
目立った周期性は無さそうです。

このように、なかなか興味深いデータが揃っているように思います。
(c) HoloViews + bokeh 使用
最後に、HoloViews + bokeh でインタラクティブに操作できるグラフを作成します。
とりあえず、感染症(列)毎に折れ線グラフ(Curve)を作って Overlay で重ねてみました。
opts でグラフのサイズやフォントサイズを変更しています。
なお、Curve で label = c とすると (label の値は凡例に使われる)、正常に動作しなかったため、回避措置として label = f"'{c}'" のようにしています。
idwr_plot_holoviews.py
import sys import pandas as pd import holoviews as hv hv.extension('bokeh') data_file = sys.argv[1] dest_file = sys.argv[2] df = pd.read_csv(data_file, parse_dates = ['lastdate']) dg = df.groupby('lastdate').sum().iloc[:, 1:20] # 感染症毎に Curve を作成 plist = [hv.Curve(dg[c].reset_index().values, label = f"'{c}'") for c in dg] # 複数の Curve を重ねる p = hv.Overlay(plist) # グラフのサイズを変更 p = p.opts(plot = dict(width = 800, height = 600, fontsize = 8)) # X軸・Y軸のラベルを変更 p = p.redim.label(x = 'lastdate', y = 'num') # グラフの保存 hv.renderer('bokeh').save(p, dest_file)
実行結果は以下の通り。
拡張子 .html は勝手に付与されるようです。
実行例
> python idwr_plot_holoviews.py idwr.csv idwr_holoviews
idwr_holoviews.html 表示例

Python でアソシエーション分析 - Orange3-Associate
前回 と同様のアソシエーション分析を Python の Orange で試してみました。
ソースは http://github.com/fits/try_samples/tree/master/blog/20180109/
はじめに
データセット
前回 と同じデータファイルを使います。
data.basket
C,S,M,R T,Y,C P,Y,C,M O,W,L R O,U,R,L P W,C T,O,W,B,C C,T,W,B,Y,F,D ・・・ R,P,S B, B,S B,F C,F,N
インストール
今回は Orange3 を使います。
Orange3 ではアソシエーション分析に関する処理を Orange3-Associate へ分離しているようなので、これらをインストールしておきます。
Orange3 インストール
Orange3 自体は conda コマンドでインストールできるようです。
Orange3 インストール例
> conda install orange3
より新しいバージョンをインストールするには conda-forge を使えば良さそうです。
Orange3 インストール例(conda-forge 利用)
> conda config --add channels conda-forge > conda install orange3
Orange3-Associate インストール
試した時点では、Orange3-Associate を conda や pip でインストールできなかったので、ソースを取得してインストールしました。
Orange3-Associate インストール例
> git clone https://github.com/biolab/orange3-associate.git > cd orange3-associate > python setup.py install
実装と実行
前回 と同様の処理を実装してみます。
(a) リフト値なし
まずは、Orange.data.Table でデータファイルを読み込みます。 (ファイル名の拡張子は .basket とする必要がありそう)
その結果、tbl 変数の内容は [[C=1.000, S=1.000, M=1.000, R=1.000], [C=1.000, T=1.000, Y=1.000], ・・・] のようになります。
C や S のような文字列では処理できないようなので、OneHot.encode で One hot 表現化します。
その結果、X 変数の内容は [[0, 1, 2, 3], [0, 4, 5], ・・・] のようになります。(出現した順に 0 からの連番が割り当てられるようです)
frequent_itemsets で組み合わせ毎の発生件数をカウントします。
第 2引数の min_support で抽出する support(支持度)の最小値を指定できます。以下のサンプルでは 5 と指定しているので 5件以上のものが抽出されます。(比率の指定も可能)
itemsets 変数の内容は {frozenset({11}): 42, frozenset({0}): 41, frozenset({0, 11}): 12, ・・・} のようになります。
association_rules でアソシエーションルールの抽出を行います。
第 2引数の min_confidence で抽出する confidence(確信度)の最小値を指定できます。
ただし、association_rules ではリフト値を取得できないようです。
P や Q 変数の内容は frozenset({11, 7}) のようになるので、元の文字列へ戻すために OneHot.decode で処理します。
OneHot.decode の結果は [(11, ContinuousVariable(name='B', number_of_decimals=3), 0), (7, ContinuousVariable(name='O', number_of_decimals=3), 0)] のようになるので ContinuousVariable の name の値を取り出しています。
sample.py
import sys import Orange from orangecontrib.associate.fpgrowth import * data_file = sys.argv[1] # データファイル読み込み tbl = Orange.data.Table(data_file) X, mapping = OneHot.encode(tbl) itemsets = dict(frequent_itemsets(X, 5)) # アソシエーションルールの抽出 rules = association_rules(itemsets, 0.7) def decode_onehot(d): items = OneHot.decode(d, tbl, mapping) # ContinuousVariable の name 値を取得 return list(map(lambda v: v[1].name, items)) for P, Q, support, confidence in rules: lhs = decode_onehot(P) rhs = decode_onehot(Q) print(f"lhs = {lhs}, rhs = {rhs}, support = {support}, confidence = {confidence}")
実行結果は以下の通り。
実行結果
> python sample.py data.basket lhs = ['B', 'O'], rhs = ['W'], support = 5, confidence = 0.8333333333333334 lhs = ['B', 'T'], rhs = ['C'], support = 5, confidence = 1.0 lhs = ['N'], rhs = ['C'], support = 10, confidence = 0.7142857142857143 lhs = ['T'], rhs = ['C'], support = 8, confidence = 0.8
(b) リフト値あり
リフト値の取得には rules_stats を使います。
rules_stats の第 3引数にはデータセットの件数(今回は 100)を指定します。(この値はリフト値の算出に使われる)
sample2.py
import sys import Orange from orangecontrib.associate.fpgrowth import * data_file = sys.argv[1] tbl = Orange.data.Table(data_file) X, mapping = OneHot.encode(tbl) itemsets = dict(frequent_itemsets(X, 5)) # アソシエーションルールの抽出 rules = association_rules(itemsets, 0.7) # リフト値を含んだ結果を取得 stats = rules_stats(rules, itemsets, len(X)) def decode_onehot(d): items = OneHot.decode(d, tbl, mapping) return list(map(lambda v: v[1].name, items)) # リフト値(7番目の要素)でソート for s in sorted(stats, key = lambda x: x[6], reverse = True): lhs = decode_onehot(s[0]) rhs = decode_onehot(s[1]) support = s[2] confidence = s[3] lift = s[6] print(f"lhs = {lhs}, rhs = {rhs}, support = {support}, confidence = {confidence}, lift = {lift}")
実行結果は以下の通り。
R の arules を使った 前回 と概ね同じ結果になりました。
実行結果
> python sample2.py data.basket lhs = ['B', 'O'], rhs = ['W'], support = 5, confidence = 0.8333333333333334, lift = 3.333333333333334 lhs = ['B', 'T'], rhs = ['C'], support = 5, confidence = 1.0, lift = 2.4390243902439024 lhs = ['T'], rhs = ['C'], support = 8, confidence = 0.8, lift = 1.951219512195122 lhs = ['N'], rhs = ['C'], support = 10, confidence = 0.7142857142857143, lift = 1.7421602787456447
MXNet で iris を分類
MXNet を使って、階層型ニューラルネットによる iris の分類を試してみました。
ソースは http://github.com/fits/try_samples/tree/master/blog/20170821/
準備
MXNet は様々なプログラミング言語用の Docker イメージを提供しているので、今回は Python 用の mxnet/python をそのまま使います。
Docker イメージの pull
$ docker pull mxnet/python
(1) iris データセット
「Keras で iris を分類」 では sklearn の iris データセットを使いましたが、今回は numpy を使って iris.data ファイルを処理してデータセットを作成します。
iris データセット (iris.data) は https://archive.ics.uci.edu/ml/datasets/iris からダウンロードし配置(今回は /vagrant/work)しておきます。
品種(Iris-setosa 等)は質的データ(カテゴリカルデータ)ですが、このままだと MXNet では扱えなさそうだったので数値化するようにしました。
numpy で該当するような関数が見当たらなかったので、品種をリスト化して該当する要素のインデックスを取得する方法で数値化するようにしてみました。 (map 処理も無さそうだったので vectorize で代用しています)
iris データセットの作成処理
import numpy as np # 品種の数値化(Iris-setosa -> 0, Iris-versicolor -> 1, Iris-virginica -> 2) def categorical(ds): ct = np.unique(ds).tolist() return np.vectorize(lambda x: ct.index(x))(ds) iris = np.loadtxt('iris.data', delimiter = ',', dtype = [ ('sepal-length', 'f4'), ('sepal-width', 'f4'), ('petal-length', 'f4'), ('petal-width', 'f4'), ('species', 'S15') ]) iris_data = np.c_[ iris['sepal-length'], iris['sepal-width'], iris['petal-length'], iris['petal-width'] ] iris_label = categorical(iris['species'])
これで iris_data と iris_label は以下のような内容になります。
iris_data の内容
[[ 5.0999999 3.5 1.39999998 0.2 ] [ 4.9000001 3. 1.39999998 0.2 ] [ 4.69999981 3.20000005 1.29999995 0.2 ] [ 4.5999999 3.0999999 1.5 0.2 ] ・・・ ・・・]
iris_label の内容
[0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 1 2 2 2 2 2 2 2 2 2 2 2 2 2 2 2 2 2 2 2 2 2 2 2 2 2 2 2 2 2 2 2 2 2 2 2 2 2 2 2 2 2 2 2 2 2 2 2 2 2 2]
(2) 学習と評価
それでは iris データセットを学習用と評価用に分割して学習と評価をそれぞれ実行してみます。
MXNet では学習や評価に使うデータは mxnet.io.DataIter で表現し、numpy の ndarray を使う場合は mx.io.NDArrayIter を使えば良さそうです。
学習・評価用のデータ分割してくれるような mxnet.io.DataIter は見当たらなかったので、「Keras で iris を分類」 と同様に random.permutation で 0 ~ 149 の数値をランダムに配置した配列を作成して、学習・評価用にデータ分割しました。
また、MXNet には以下のような API が用意されていますが、今回は Module API の方を使う事にします。
Module API では mx.sym の FullyConnected 等を使ってニューラルネットの構成を定義して mx.mod.Module でモジュールを作成、fit で学習、score で評価を実施できます。
/vagrant/work/iris_sample.py
import mxnet as mx import numpy as np train_test_rate = 0.7 def categorical(ds): ct = np.unique(ds).tolist() return np.vectorize(lambda x: ct.index(x))(ds) iris = np.loadtxt('iris.data', delimiter = ',', dtype = [ ('sepal-length', 'f4'), ('sepal-width', 'f4'), ('petal-length', 'f4'), ('petal-width', 'f4'), ('species', 'S15') ]) iris_data = np.c_[ iris['sepal-length'], iris['sepal-width'], iris['petal-length'], iris['petal-width'] ] iris_label = categorical(iris['species']) data_size = len(iris) train_size = int(data_size * train_test_rate) perm = np.random.permutation(data_size) train_perm = perm[0:train_size] test_perm = perm[train_size:] # 学習用データセット train_iter = mx.io.NDArrayIter(iris_data[train_perm], iris_label[train_perm]) # 評価用データセット test_iter = mx.io.NDArrayIter(iris_data[test_perm], iris_label[test_perm]) # ニューラルネットの構成定義 data = mx.sym.Variable('data') net = mx.sym.FullyConnected(data = data, name = 'fc1', num_hidden = 5) net = mx.sym.Activation(data = net, name = 'relu1', act_type = 'relu') net = mx.sym.FullyConnected(data = net, name = 'fc2', num_hidden = 3) net = mx.sym.SoftmaxOutput(data = net, name = 'softmax') # モジュールの作成 mod = mx.mod.Module(net) # 学習 mod.fit(train_iter, num_epoch = 20) # 評価 res = mod.score(test_iter, mx.metric.Accuracy()) print(res)
mxnet/python の Docker コンテナを起動して実行します。
実行例
$ docker run -it --rm -v /vagrant/work:/work mxnet/python
・・・# cd /work
・・・# python iris_sample.py
[('accuracy', 0.9555555555555556)]
Python で HAR ファイルから散布図を作成
Web サイトの構成を可視化するため、Python で HAR (HTTP ARchive) ファイルをパースし散布図を試しに作ってみました。
今回作成した散布図の内容は以下の通りです。
- 横軸は time (処理時間 (ミリ秒))
- 縦軸は bodySize (レスポンスボディのサイズ)
- MIME タイプのサブタイプ別に色分け
今回は Anaconda に予め含まれているライブラリを使用する事にします。
今回のソースは http://github.com/fits/try_samples/tree/master/blog/20170514/
はじめに
HAR ファイルは Chrome や Firefox の開発ツールで取得できますが、簡易なものは PhantomJS の examples/netsniff.js で取得できるので今回はこれを使いました。
HAR の取得
phantomjs netsniff.js <URL>
netsniff.js では console.log で HAR の内容を出力するようになっています。
HAR ファイルの取得例
phantomjs netsniff.js https://www.・・・ > a.har
HAR ファイルは以下のような JSON 形式のファイルとなっています。
HAR ファイル例
{ "log": { ・・・ "entries": [ { ・・・ "time": 4474, "request": { ・・・ }, "response": { ・・・ "bodySize": 473, "content": { "size": 473, "mimeType": "text/html;charset=UTF-8" } }, "cache": {}, "timings": { ・・・ "wait": 3813, "receive": 661, "ssl": -1 }, ・・・ }, ・・・ ] } }
netsniff.js では、timings 内の wait と receive 以外の値は固定値(0 か -1)が設定されるようになっています。
1. seaborn の pairplot 使用
まずは、seaborn の pairplot を使って散布図を描画してみます。 (pairplot はデフォルトが散布図になっているようです)
pairplot は基本的にデータ内の数値変数の全ての組み合わせをグラフ化するものですが、x_vars と y_vars を指定する事で特定の組み合わせに限定したグラフだけを描画する事も可能です。
色分けする項目は hue で指定できるようになっています。
複数のグラフをグリッド表示する事を想定しているため、個々のグラフのデフォルトサイズが小さく設定されていますが、size で変更する事が可能です。
har_scatter_plot1.py
import sys import json import codecs import pandas as pd import seaborn as sns import matplotlib.pyplot as plt # HAR ファイルの読み込みと Python オブジェクト化 data = json.load(codecs.open(sys.argv[1], 'r', 'utf-8')) to_subtype = lambda mtype: mtype.split(';')[0].split('/')[-1] # 特定の項目を抜き出す処理 convert = lambda d: { 'subType': to_subtype(d['response']['content']['mimeType']), 'bodySize': d['response']['bodySize'], 'time': d['time'] } # DataFrame 化 df = pd.DataFrame(list(map(convert, data['log']['entries']))) # 散布図の描画 sns.pairplot(df, hue = 'subType', x_vars = 'time', y_vars = 'bodySize', size = 10) imgfile = "%s_1.png" % sys.argv[1] # グラフの保存 plt.savefig(imgfile)
実行例
python har_scatter_plot1.py a.har
実行結果例
a.har_1.png

b.har_1.png

2. サブタイプ毎に plot
pairplot の結果ではサブタイプ毎の色が固定されておらず、複数の Web ページを比較する用途には適していません。
そこで、サブタイプ毎の散布図を重ねて描画してみました。
har_scatter_plot2.py
import sys import json import codecs import pandas as pd import matplotlib.pyplot as plt ・・・ # サブタイプと色のマッピング color_map = { 'javascript': 'blue', 'x-javascript': 'blue', 'json': 'green', 'gif': 'tomato', 'jpeg': 'red', 'png': 'pink', 'html': 'lime', 'css': 'turquoise' } df = pd.DataFrame(list(map(convert, data['log']['entries']))) ax = plt.figure().add_subplot(1, 1, 1) for (k, c) in color_map.items(): # 指定サブタイプのデータを抽出 sdf = df[df['subType'] == k] if not sdf.empty: # c の色で散布図(scatter)を描画 sdf.plot('time', 'bodySize', 'scatter', ax, c = c, label = k) imgfile = "%s_2.png" % sys.argv[1] plt.savefig(imgfile)
実行例
python har_scatter_plot2.py a.har
実行結果例
a.har_2.png

b.har_2.png
