Gradle を使った Querydsl SQL のコード生成
前々回 と 前回 に続き、今回は Querydsl SQL のコード生成を Gradle で実施してみました。
ソースは http://github.com/fits/try_samples/tree/master/blog/20150413/
なお、Querydsl 4.0 からパッケージ名等が変更になるようなのでご注意ください。(github の master ブランチでは com.querydsl となっています)
Gradle を使ったコード生成
Querydsl SQL の場合は Querydsl JPA・MongoDB と違って DB からメタデータを取得してコードを生成します。
querydsl-sql-codegen モジュールを使用し、アノテーションプロセッサではなく Ant 用のタスクや API を使ってコードを生成します。
(a) Ant 用のタスクを使用
まずは Ant 用のタスククラス com.mysema.query.sql.ant.AntMetaDataExporter を Gradle から使ってコード生成してみます。
基本的に、DB への接続が発生するため JDBC ドライバーが必要となります。 また、コード生成のログを出力するには slf4j を使います。(slf4j を使わなくても支障はありません)
今回は MySQL を使いました。
build1.gradle
apply plugin: 'java' // DB 接続 URL def qdslDbUrl = 'jdbc:mysql://localhost:3306/sample1?user=root' // コード生成先ディレクトリ def qdslDestDir = 'src/main/qdsl-generated' // 生成するコードの所属パッケージ def qdslDestPackage = 'sample.model' repositories { jcenter() } configurations { querydsl } dependencies { // コード生成用の依存モジュール定義 querydsl 'com.mysema.querydsl:querydsl-sql-codegen:3.6.3' querydsl 'mysql:mysql-connector-java:5.1.35' querydsl 'org.slf4j:slf4j-simple:1.7.12' compile 'com.mysema.querydsl:querydsl-sql:3.6.3' } task generate << { // Ant 用のタスク定義 ant.taskdef( name: 'export', classname: 'com.mysema.query.sql.ant.AntMetaDataExporter', classpath: configurations.querydsl.asPath ) // コード生成の実行 ant.export( jdbcDriverClass: 'com.mysql.jdbc.Driver', dbUrl: qdslDbUrl, targetSourceFolder: qdslDestDir, targetPackage: qdslDestPackage ) } compileJava { dependsOn generate sourceSets.main.java.srcDir qdslDestDir } clean { delete qdslDestDir }
ビルド結果は以下の通りです。
事前に MySQL を起動し、テーブル等を作成しておきます。
ビルド例
> gradle -b build1.gradle build :generate [ant:export] [main] INFO com.mysema.query.sql.codegen.MetaDataExporter - Exported product successfully [ant:export] [main] INFO com.mysema.query.sql.codegen.MetaDataExporter - Exported variation successfully :compileJava :processResources UP-TO-DATE :classes :jar :assemble :compileTestJava UP-TO-DATE :processTestResources UP-TO-DATE :testClasses UP-TO-DATE :test UP-TO-DATE :check UP-TO-DATE :build BUILD SUCCESSFUL
generate タスクの実行時に以下のようなソースコードが生成されました。
- src/main/qdsl-generated/sample/model/QProduct.java
- src/main/qdsl-generated/sample/model/QVariation.java
ちなみに、今回は以下のようなテーブルを用意しています。
DDL 例
CREATE TABLE `product` ( `id` int(11) NOT NULL AUTO_INCREMENT, `name` varchar(50) NOT NULL, PRIMARY KEY (`id`) ) ENGINE=InnoDB DEFAULT CHARSET=utf8; CREATE TABLE `variation` ( `product_id` int(11) NOT NULL, `size` varchar(10) NOT NULL, `color` varchar(10) NOT NULL, PRIMARY KEY (`product_id`,`size`,`color`) ) ENGINE=InnoDB DEFAULT CHARSET=utf8;
(b) API を使用
次は、コード生成の API (com.mysema.query.sql.codegen.MetaDataExporter クラス)を Gradle から直接使います。
MetaDataExporter クラスへ出力パッケージや出力ディレクトリなどを設定した後、export メソッドへ DatabaseMetaData を渡せばコードを生成します。
なお、今回は使っていませんが、com.mysema.query.sql.Configuration で特定のカラムの型 (コード生成の Java の型) を変更するような事も可能です。
build2.gradle
apply plugin: 'java' def qdslDbUrl = 'jdbc:mysql://localhost:3306/sample1?user=root' def qdslDestDir = 'src/main/qdsl-generated' def qdslDestPackage = 'sample.model' buildscript { repositories { jcenter() } dependencies { // コード生成用の依存モジュール定義 classpath 'com.mysema.querydsl:querydsl-sql-codegen:3.6.3' classpath 'mysql:mysql-connector-java:5.1.35' } } repositories { jcenter() } dependencies { compile 'com.mysema.querydsl:querydsl-sql:3.6.3' } task generate << { // DB 接続 def con = new com.mysql.jdbc.Driver().connect(qdslDbUrl, null) def exporter = new com.mysema.query.sql.codegen.MetaDataExporter() exporter.targetFolder = new File(qdslDestDir) exporter.packageName = qdslDestPackage // コード生成の実行 exporter.export(con.metaData) con.close() } compileJava { dependsOn generate sourceSets.main.java.srcDir qdslDestDir } clean { delete qdslDestDir }
ビルド結果は以下の通りです。
ビルド例
> gradle -b build2.gradle build :generate :compileJava :processResources UP-TO-DATE :classes :jar :assemble :compileTestJava UP-TO-DATE :processTestResources UP-TO-DATE :testClasses UP-TO-DATE :test UP-TO-DATE :check UP-TO-DATE :build BUILD SUCCESSFUL
成果物は (a) のケースと同じです。
サンプルアプリケーション
最後に、簡単なサンプルアプリケーションを実装し実行してみます。
ビルド定義
(b) のビルド定義をベースに以下のようなビルド定義を用意しました。
build.gradle
apply plugin: 'application' def enc = 'UTF-8' tasks.withType(AbstractCompile)*.options*.encoding = enc def qdslDbUrl = 'jdbc:mysql://localhost:3306/sample1?user=root' def qdslDestDir = 'src/main/qdsl-generated' def qdslDestPackage = 'sample.model' // 実行クラス mainClassName = 'sample.App' buildscript { repositories { jcenter() } dependencies { classpath 'com.mysema.querydsl:querydsl-sql-codegen:3.6.3' classpath 'mysql:mysql-connector-java:5.1.35' } } repositories { jcenter() } dependencies { compile 'com.mysema.querydsl:querydsl-sql:3.6.3' // 実行用の依存モジュール定義 runtime 'mysql:mysql-connector-java:5.1.35' runtime 'org.slf4j:slf4j-nop:1.7.12' } task generate << { def con = new com.mysql.jdbc.Driver().connect(qdslDbUrl, null) def exporter = new com.mysema.query.sql.codegen.MetaDataExporter() exporter.targetFolder = new File(qdslDestDir) exporter.packageName = qdslDestPackage exporter.export(con.metaData) con.close() } compileJava { dependsOn generate sourceSets.main.java.srcDir qdslDestDir } clean { delete qdslDestDir }
アプリケーションクラス
product・variation テーブルへそれぞれデータを insert して select するだけの単純な処理を実装しました。
insert は set メソッドを使う方法と columns・values メソッドを使う方法を用いてみました。 (他にも Bean を populate する方法があります)
src/main/sample/App.java
package sample; import com.mysema.query.sql.Configuration; import com.mysema.query.sql.MySQLTemplates; import com.mysema.query.sql.SQLQuery; import com.mysema.query.sql.dml.SQLInsertClause; import sample.model.QProduct; import sample.model.QVariation; import java.sql.Connection; import java.sql.DriverManager; import java.sql.SQLException; public class App { public static void main(String... args) throws SQLException { String dbUrl = "jdbc:mysql://localhost:3306/sample1?user=root"; Configuration conf = new Configuration(new MySQLTemplates()); QProduct p = QProduct.product; QVariation v = QVariation.variation; try (Connection con = DriverManager.getConnection(dbUrl)) { con.setAutoCommit(false); // product テーブルへレコードを insert Integer id = new SQLInsertClause(con, conf, p) .set(p.name, "test" + System.currentTimeMillis()) .executeWithKey(p.id); // variation テーブルへレコードを insert new SQLInsertClause(con, conf, v) .columns(v.productId, v.size, v.color) .values(id, "L", "white") .execute(); // variation テーブルへレコードを insert new SQLInsertClause(con, conf, v) .columns(v.productId, v.size, v.color) .values(id, "S", "blue") .execute(); con.commit(); // product と variation を join して select new SQLQuery(con, conf) .from(p) .join(v).on(v.productId.eq(p.id)) .where(p.name.startsWith("test")) .list(p.id, p.name, v.color, v.size) .forEach(System.out::println); } } }
実行
実行結果は以下の通りです。
実行結果
> gradle run ・・・ :run [1, test1428773665876, white, L] [1, test1428773665876, blue, S] BUILD SUCCESSFUL
Gradle を使った Querydsl MongoDB のコード生成
前回の Querydsl JPA に続き、今回は Querydsl MongoDB のコード生成を Gradle で実施してみました。
ソースは http://github.com/fits/try_samples/tree/master/blog/20150330/
Gradle を使ったコード生成
Querydsl MongoDB の場合は、前回 のビルド定義ファイルから依存モジュールとアノテーションプロセッサクラスを以下のように変えるだけです。 (querydsl-apt はそのまま使います)
- querydsl-jpa を
querydsl-mongodbへ変更 - javaee-api を
morphiaへ変更 - com.mysema.query.apt.jpa.JPAAnnotationProcessor を
com.mysema.query.apt.morphia.MorphiaAnnotationProcessorへ変更
なお、エンティティクラスは Morphia のアノテーションを使って定義します。
(a) compileJava タスクでコード生成とコンパイルを実施
compileJava タスクの実行時に Querydsl のコード生成とコンパイルの両方を実施するタイプです。
build1.gradle
apply plugin: 'java' repositories { jcenter() } configurations { apt } dependencies { apt 'com.mysema.querydsl:querydsl-apt:3.6.2' // Querydsl MongoDB 用の依存モジュール compile 'com.mysema.querydsl:querydsl-mongodb:3.6.2' compile 'org.mongodb.morphia:morphia:0.110' } compileJava { classpath += configurations.apt options.compilerArgs += [ '-processor', 'com.mysema.query.apt.morphia.MorphiaAnnotationProcessor' ] } jar { excludes << '**/*.java' }
ビルド結果は以下の通りです。
ビルド例
> gradle -b build1.gradle build :compileJava 注意:Running MorphiaAnnotationProcessor 注意:Serializing Entity types 注意:Generating sample.model.QProduct for [sample.model.Product] 注意:Serializing Embeddable types 注意:Generating sample.model.QVariation for [sample.model.Variation] 注意:Running MorphiaAnnotationProcessor 注意:Running MorphiaAnnotationProcessor :processResources UP-TO-DATE :classes :jar :assemble :compileTestJava UP-TO-DATE :processTestResources UP-TO-DATE :testClasses UP-TO-DATE :test UP-TO-DATE :check UP-TO-DATE :build BUILD SUCCESSFUL
自動生成されるソースの場所やクラス名の先頭に Q が付くのは前回と同じです。
(b) 別タスクでコード生成
Querydsl のコード生成を別タスク (下記の generate) で実施するタイプです。
Querydsl は IDE で利用すると思いますので、こちらの方が使い易いと思います。
build2.gradle
apply plugin: 'java' def qdslDestDir = 'src/main/qdsl-generated' repositories { jcenter() } configurations { apt } dependencies { apt 'com.mysema.querydsl:querydsl-apt:3.6.2' compile 'com.mysema.querydsl:querydsl-mongodb:3.6.2' compile 'org.mongodb.morphia:morphia:0.110' } task generate(type: JavaCompile) { source = sourceSets.main.java classpath = configurations.compile + configurations.apt destinationDir = new File(qdslDestDir) options.compilerArgs += [ '-proc:only', '-processor', 'com.mysema.query.apt.morphia.MorphiaAnnotationProcessor' ] } compileJava { dependsOn generate sourceSets.main.java.srcDir qdslDestDir } clean { delete qdslDestDir }
ビルド結果は以下の通りです。
ビルド例
> gradle -b build2.gradle build :generate 注意:Running MorphiaAnnotationProcessor 注意:Serializing Entity types 注意:Generating sample.model.QProduct for [sample.model.Product] 注意:Serializing Embeddable types 注意:Generating sample.model.QVariation for [sample.model.Variation] 注意:Running MorphiaAnnotationProcessor 注意:Running MorphiaAnnotationProcessor :compileJava :processResources UP-TO-DATE :classes :jar :assemble :compileTestJava UP-TO-DATE :processTestResources UP-TO-DATE :testClasses UP-TO-DATE :test UP-TO-DATE :check UP-TO-DATE :build BUILD SUCCESSFUL
サンプルアプリケーション
最後に、簡単なサンプルアプリケーションを実装し実行してみます。
ビルド定義
別タスクによるコード生成タイプ (b) のビルド定義をベースに以下のようなビルド定義を用意しました。
build.gradle
apply plugin: 'application' def enc = 'UTF-8' tasks.withType(AbstractCompile)*.options*.encoding = enc def qdslDestDir = 'src/main/qdsl-generated' repositories { jcenter() } configurations { apt } dependencies { apt 'com.mysema.querydsl:querydsl-apt:3.6.2' compile 'com.mysema.querydsl:querydsl-mongodb:3.6.2' compile 'org.mongodb.morphia:morphia:0.110' } task generate(type: JavaCompile) { source = sourceSets.main.java classpath = configurations.compile + configurations.apt destinationDir = new File(qdslDestDir) options.compilerArgs += [ '-proc:only', '-processor', 'com.mysema.query.apt.morphia.MorphiaAnnotationProcessor' ] } compileJava { dependsOn generate sourceSets.main.java.srcDir qdslDestDir } clean { delete qdslDestDir } mainClassName = 'sample.App'
エンティティクラス
Morphia 用のエンティティクラスとして以下を定義しました。
src/main/sample/model/Product.java
package sample.model; import org.mongodb.morphia.annotations.*; import org.bson.types.ObjectId; import java.util.ArrayList; import java.util.List; @Entity public class Product { @Id private ObjectId id; private String name; @Embedded private List<Variation> variationList = new ArrayList<>(); public Product() { } public Product(String name) { setName(name); } public void setId(ObjectId id) { this.id = id; } public ObjectId getId() { return id; } public void setName(String name) { this.name = name; } public String getName() { return name; } public List<Variation> getVariationList() { return variationList; } }
src/main/sample/model/Variation.java
package sample.model; import org.mongodb.morphia.annotations.Embedded; @Embedded public class Variation { private String size; private String color; public Variation() { } public Variation(String size, String color) { setSize(size); setColor(color); } public String getSize() { return size; } public void setSize(String size) { this.size = size; } public String getColor() { return color; } public void setColor(String color) { this.color = color; } }
アプリケーションクラス
Product を保存した後に、Querydsl MongoDB の機能で簡単な検索を行う処理を実装しました。
src/main/sample/App.java
package sample; import com.mongodb.MongoClient; import com.mysema.query.mongodb.morphia.MorphiaQuery; import org.mongodb.morphia.Datastore; import org.mongodb.morphia.Morphia; import sample.model.Product; import sample.model.QProduct; import sample.model.Variation; import java.net.UnknownHostException; public class App { public static void main(String... args) throws UnknownHostException { Morphia morphia = new Morphia(); Datastore ds = morphia.createDatastore(new MongoClient(), "sample"); Product p = new Product("test" + System.currentTimeMillis()); p.getVariationList().add(new Variation("L", "black")); p.getVariationList().add(new Variation("M", "white")); ds.save(p); // Querydsl MongoDB を使った検索処理 QProduct qp = QProduct.product; MorphiaQuery<Product> query = new MorphiaQuery<>(morphia, ds, qp); query.where(qp.name.like("test%")).list().forEach(App::printProduct); } private static void printProduct(Product p) { System.out.println("----------"); System.out.println(p.getId() + ", " + p.getName()); p.getVariationList().forEach(v -> System.out.println(v.getColor() + ", " + v.getSize())); } }
実行
まず、MongoDB を起動しておきます。 今回は開発版の MongoDB 3.1.0 (SSL版) を使用しました。
MongoDB 起動
> mongod --dbpath data ・・・ 2015-03-29T23:50:32.137+0900 I CONTROL [initandlisten] ** NOTE: This is a development version (3.1.0) of MongoDB. ・・・ 2015-03-29T23:50:32.685+0900 I NETWORK [initandlisten] waiting for connections on port 27017
実行結果は以下の通りです。
実行結果
> gradle run ・・・ :run 3 29, 2015 11:52:47 午後 org.mongodb.morphia.logging.MorphiaLoggerFactory choose LoggerFactory 情報: LoggerImplFactory set to org.mongodb.morphia.logging.jdk.JDKLoggerFactory ---------- 551811bfcc87cb6a0dcb43fd, test1427640767733 black, L white, M BUILD SUCCESSFUL
MongoDB shell を使って登録内容を確認してみます。
データ確認
> mongo
・・・
MongoDB shell version: 3.1.0
connecting to: test
Welcome to the MongoDB shell.
・・・
> use sample
switched to db sample
> db.Product.find()
{ "_id" : ObjectId("551811bfcc87cb6a0dcb43fd"), "className" : "sample.model.Product", "name" : "test1427640767733", "variationList" : [ { "size" : "L", "color"
: "black" }, { "size" : "M", "color" : "white" } ] }
Gradle を使った Querydsl JPA のコード生成
今回は Querydsl JPA のコード生成を Gradle で実施してみました。
ソースは http://github.com/fits/try_samples/tree/master/blog/20150322/
はじめに
Querydsl JPA では JPA のエンティティクラスを元に Querydsl JPA 用のコードを自動生成して使います。
この場合のコード生成は querydsl-apt モジュールに含まれているアノテーションプロセッサ com.querydsl.apt.jpa.JPAAnnotationProcessor を javac のコンパイルオプションへ指定するだけです。
javac -processor com.mysema.query.apt.jpa.JPAAnnotationProcessor ・・・
Gradle の場合でも同様に上記コンパイルオプションを使用します。
(a) compileJava タスクでコード生成とコンパイルを実施
まずは、compileJava タスクの実行時に Querydsl のコード生成とコンパイルの両方を実施してみます。
下記 (1) のように compileJava の options.compilerArgs へコンパイルオプション -processor com.mysema.query.apt.jpa.JPAAnnotationProcessor を追加します。
こうすることで、src/main/java 内の JPA エンティティクラスを元に destinationDir プロパティ値のディレクトリ (デフォルトでは build/classes/main) へ Querydsl JPA 用のソースファイルを自動生成し、コンパイルを実施します。
デフォルトでは destinationDir 内の全ファイルを JAR ファイルへ格納してしまいますので、下記の (2) では jar タスクの excludes を使って Querydsl が生成したソースファイルを除外するようにしています。
build.gradle
apply plugin: 'java' repositories { jcenter() } configurations { apt } dependencies { // Querydsl JPA のコード生成モジュール apt 'com.mysema.querydsl:querydsl-apt:3.6.2' compile 'com.mysema.querydsl:querydsl-jpa:3.6.2' compile 'javax:javaee-api:7.0' } compileJava { classpath += configurations.apt // (1) アノテーションプロセッサの利用設定 options.compilerArgs += [ '-processor', 'com.mysema.query.apt.jpa.JPAAnnotationProcessor' ] } jar { // (2) 自動生成したソースファイルを jar ファイルから除外するための設定 excludes << '**/*.java' }
ビルド結果は以下の通りです。
ビルド例
> gradle build :compileJava 注意:Running JPAAnnotationProcessor 注意:Serializing Entity types 注意:Generating sample.model.QProduct for [sample.model.Product] 注意:Running JPAAnnotationProcessor 注意:Running JPAAnnotationProcessor :processResources UP-TO-DATE :classes :jar :assemble :compileTestJava UP-TO-DATE :processTestResources UP-TO-DATE :testClasses UP-TO-DATE :test UP-TO-DATE :check UP-TO-DATE :build BUILD SUCCESSFUL
JPA 用のエンティティクラス src/main/java/sample/model/Product.java を元に build/classes/main/sample/model/QProduct.java ファイルを生成し、ビルドを行っています。
ビルド結果の JAR ファイルは以下のような内容になります。
JAR ファイルの内容例
- sample/model
- Product.class
- QProduct.class
- META-INF
- MANIFEST.MF
(b) 別タスクでコード生成
次は、Querydsl のコード生成を別タスク (下記の generate) で実施するようにしてみます。
javac のコンパイルオプションで -proc:only とすれば、コンパイルを実施せずアノテーションプロセッサだけを実施するようになりますので、これを利用します。
JavaCompile を type へ指定したタスクを定義して source・classpath・destinationDir (コード生成の出力先) を設定し、コンパイルオプションへ -proc:only を追加します。
あとは compileJava タスクのコンパイル対象ソースへ Querydsl コード生成先ディレクトリを追加するだけです。 (タスクの依存設定は必要に応じて実施)
build2.gradle
apply plugin: 'java' // コード生成先 def qdslDestDir = 'src/main/qdsl-generated' repositories { jcenter() } configurations { apt } dependencies { apt 'com.mysema.querydsl:querydsl-apt:3.6.2' compile 'com.mysema.querydsl:querydsl-jpa:3.6.2' compile 'javax:javaee-api:7.0' } // Querydsl JPA コード生成用のタスク定義 task generate(type: JavaCompile) { source = sourceSets.main.java classpath = configurations.compile + configurations.apt // コード生成の出力先を設定 destinationDir = new File(qdslDestDir) options.compilerArgs += [ '-proc:only', // アノテーションプロセッサのみ実施するための設定 (コンパイルしない) '-processor', 'com.mysema.query.apt.jpa.JPAAnnotationProcessor' ] } compileJava { dependsOn generate // コンパイル対象ソースへコード生成先ディレクトリを追加 sourceSets.main.java.srcDir qdslDestDir } clean { delete qdslDestDir }
ビルド結果は以下の通りです。
アノテーションプロセッサを compileJava タスクの前に実行している以外は先程の (a) と同じです。 (当然ながら JAR ファイルの内容も同じです)
ビルド例
> gradle -b build2.gradle build :generate 注意:Running JPAAnnotationProcessor 注意:Serializing Entity types 注意:Generating sample.model.QProduct for [sample.model.Product] 注意:Running JPAAnnotationProcessor 注意:Running JPAAnnotationProcessor :compileJava :processResources UP-TO-DATE :classes :jar :assemble :compileTestJava UP-TO-DATE :processTestResources UP-TO-DATE :testClasses UP-TO-DATE :test UP-TO-DATE :check UP-TO-DATE :build BUILD SUCCESSFUL
Julia でロジスティック回帰 - glm
前回 に続き、今回も Julia で GLM を実施します。
今回は 「R でロジスティック回帰 - glm, MCMCpack」 のロジスティック回帰(GLM)を Julia で実装してみました。
サンプルソースは http://github.com/fits/try_samples/tree/master/blog/20150309/
はじめに
GLM 等のパッケージは 前回 と同じものを使用します。
データは R で試した時のものをそのまま使います。(「R でロジスティック回帰 - glm, MCMCpack」 参照)
データ data4a.csv
N,y,x,f 8,1,9.76,C 8,6,10.48,C 8,5,10.83,C ・・・
データの内容は以下の通りで、個体 i それぞれにおいて 「 個の観察種子のうち生きていて発芽能力があるものは
個」 となっています。
| 項目 | 内容 |
|---|---|
| N | 観察種子数 |
| y | 生存種子数 |
| x | 植物の体サイズ |
| f | 施肥処理 (C: 肥料なし, T: 肥料あり) |
体サイズ x と肥料による施肥処理 f が種子の生存する確率(ある個体 i から得られた種子が生存している確率)にどのように影響しているかをロジスティック回帰で解析します。
GLM によるロジスティック回帰
Julia の GLM パッケージを使う場合の注意点は下記の通りです。
- (1) Binomial (二項分布) を使う場合は応答変数(下記の y に該当)の値が 0.0 ~ 1.0 内でなければならない
- (2) readtable ではカテゴリ型変数 (R の因子型に該当) 化を実施しない
(1) への対応として、y を N で割った値 yn を応答変数(目的変数)として使いました。
今のところ d[:yn] = d[:y] / d[:N] とは書けないようなので map 関数を使っています。
R の read.csv 関数等では f のような項目は因子型になると思いますが、Julia の readtable では (2) のようにカテゴリ型変数とはならず単なる文字列の配列となります。 (d[:f] の型は DataArrays.DataArray{UTF8String,1} になる)
glm 関数で文字列の変数は使えないので、カテゴリ型変数への変換が必要となります。
DataFrames パッケージにはカテゴリ型への変換関数 pool が用意されているので、下記では f 項目の値を pool 関数で変換し ff へ設定しています。 (d[:ff] の型は DataArrays.PooledDataArray{UTF8String,UInt8,1} になる)
logisticGlm.jl
using DataFrames, GLM d = readtable("data4a.csv") # (1) 生存率 y / N の算出 d[:yn] = map(x -> d[:y][x] / d[:N][x], 1:nrow(d)) # (2) カテゴリ型へ変換 (DataArrays.PooledDataArray{UTF8String,UInt8,1} になる) d[:ff] = pool(d[:f]) # 以下でも可 (ただし、DataArrays.PooledDataArray{UTF8String,UInt32,1} になる) # d[:ff] = convert(PooledDataArray, d[:f]) res = glm(yn~x + ff, d, Binomial()) println(res)
実行結果は以下のようになりました。 (R で実施したときと同じ Estimate 値)
実行結果
> julia logisticGlm.jl
DataFrames.DataFrameRegressionModel{GLM.GeneralizedLinearModel{GLM.GlmResp{Array{Float64,1},Distributions.Binomial,GLM.LogitLink},GLM.DensePredChol{Float64}},Float64}:
Coefficients:
Estimate Std.Error z value Pr(>|z|)
(Intercept) -19.5361 3.99861 -4.88572 <1e-5
x 1.95241 0.392777 4.97077 <1e-6
ff - T 2.02151 0.654152 3.09027 0.0020
ちなみに、d[:f] の内容と pool・convert 関数の適用結果は以下のようになります。
julia> d[:f]
100-element DataArrays.DataArray{UTF8String,1}:
"C"
"C"
・・・
"T"
"T"
julia> pool(d[:f])
100-element DataArrays.PooledDataArray{UTF8String,UInt8,1}:
"C"
"C"
・・・
"T"
"T"
julia> convert(PooledDataArray, d[:f])
100-element DataArrays.PooledDataArray{UTF8String,UInt32,1}:
"C"
"C"
・・・
"T"
"T"
PooledDataArray
PooledDataArray の水準名の一覧は levels 関数か pool フィールドを使って取得できます。
julia> levels(d[:ff])
2-element Array{UTF8String,1}:
"C"
"T"
julia> d[:ff].pool
2-element Array{UTF8String,1}:
"C"
"T"
水準名と数値のマッピングは levelsmap 関数で取得できます。
julia> levelsmap(d[:ff])
Dict{UTF8String,Int64} with 2 entries:
"T" => 2
"C" => 1
また、refs フィールドで各データに割り当てられた数値を取得できます。
julia> d[:ff].refs
100-element Array{UInt8,1}:
0x01
0x01
・・・
0x02
0x02
予測線の描画
次に glm の結果 (DataFrameRegressionModel) を使って予測腺を描画します。
glm の結果では、「肥料あり "T" => 1、肥料なし "C" => 0」 の扱いになっているので、predict する際に肥料ありは 1 を肥料なしは 0 の配列を渡します。
predict の結果は生存率 yn の予測値なので、グラフへ描画する際に観察種子数 N の最大値 (今回は全て 8 なので最大値とする必要はない) を乗算しています。
なお、rep 関数を使うと指定の配列を指定回数繰り返した配列を作成できます。
logisticGlm_draw.jl
using DataFrames, GLM, Gadfly ・・・ res = glm(yn~x + ff, d, Binomial()) # x の最小値 7.66 ~ 最大値 12.44 まで 0.1 刻みのデータを用意 xx = [minimum(d[:x]):0.1:maximum(d[:x])] # 肥料あり "T" の予測値を算出 rt = predict(res, DataFrame(n = rep([1], length(xx)), x = xx, ff = rep([1], length(xx)))) # 肥料なし "C" の予測値を算出 rc = predict(res, DataFrame(n = rep([1], length(xx)), x = xx, ff = rep([0], length(xx)))) p = plot( layer(d, x = "x", y = "y", color = "f", Geom.point), # 肥料あり "T" の予測線を赤で描画 layer(x = xx, y = maximum(d[:N]) * rt, Geom.line, Theme(default_color = color("red"))), # 肥料なし "C" の予測線を緑で描画 layer(x = xx, y = maximum(d[:N]) * rc, Geom.line, Theme(default_color = color("green"))) ) draw(PNG("logisticGlm_draw.png", 500px, 400px), p)
実行結果
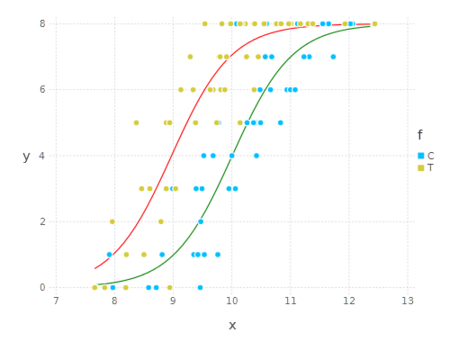
なお、rt と rc の算出処理を PooledDataArray を使って実装すると以下のようになります。
PooledDataArray では "T" => 2, "C" => 1 となっているため、"T" => 1, "C" => 0 でそれぞれ predict するように <PooledDataArray変数>.refs - 1 としています。
・・・ xx = [minimum(d[:x]):0.1:maximum(d[:x])] # 肥料あり "T" のカテゴリ型データを作成 (d[:ff] の水準を使用) ft = PooledDataArray(rep([utf8("T")], length(xx)), d[:ff].pool) # 肥料なし "C" のカテゴリ型データを作成 (d[:ff] の水準を使用) fc = PooledDataArray(rep([utf8("C")], length(xx)), d[:ff].pool) # 肥料あり "T" の予測値を算出 rt = predict(res, DataFrame(n = rep([1], length(xx)), x = xx, ff = ft.refs - 1)) # 肥料なし "C" の予測値を算出 rc = predict(res, DataFrame(n = rep([1], length(xx)), x = xx, ff = fc.refs - 1)) ・・・
Julia でポアソン回帰 - glm
以前、「R でポアソン回帰 - glm, MCMCpack」 にて試した GLM によるポアソン回帰を Julia で実施してみました。
なお、Julia は開発中の v0.4.0 を使用しました。
サンプルソースは http://github.com/fits/try_samples/tree/master/blog/20150223/
はじめに
今回は下記のパッケージを使用しますので、予めインストールしておきます。
| パッケージ名 | 用途 |
|---|---|
| DataFrames | csv ファイルの読み込み。データフレーム化 |
| GLM | GLM の実施 |
| Gadfly | グラフの描画 |
| Cairo | PNGファイルへのグラフ描画 |
パッケージのインストール例
> julia ・・・ julia> Pkg.add("DataFrames") ・・・ julia> Pkg.add("GLM") ・・・ julia> Pkg.add("Gadfly") ・・・ julia> Pkg.add("Cairo") ・・・
ポアソン回帰を試すデータは 以前 に使ったものをそのまま使用します。
データ data3a.csv
y,x,f 6,8.31,C 6,9.44,C 6,9.5,C 12,9.07,C 10,10.16,C ・・・
データ内容は下記の通りです。
今回は、体サイズ x が種子数 y にどのように影響しているかをポアソン回帰で解析します。
| 項目 | 内容 |
|---|---|
| y | 種子数 |
| x | 植物の体サイズ |
| f | 施肥処理 |
GLM によるポアソン回帰
y ~ x のモデルを使ってポアソン回帰を実施します。
以前、R で実施したものと同じポアソン回帰を行うには、glm 関数へ Poisson() と LogLink() (リンク関数)を指定します。
poissonGlm.jl
using DataFrames, GLM d = readtable("data3a.csv") res = glm(y ~ x, d, Poisson(), LogLink()) println(res)
実行結果は、以下のように R の結果とほぼ同じになりました。 (前回 の結果を参照)
今回は Julia の開発バージョンを使ったため deprecated syntax の WARNING がいくつか出力されました。
実行結果
> julia poissonGlm.jl
・・・
Coefficients:
Estimate Std.Error z value Pr(>|z|)
(Intercept) 1.29172 0.363686 3.55174 0.0004
x 0.0756619 0.0356042 2.12509 0.0336
この結果より、平均種子数の予測値を求める関数は となります。
予測線の描画1
次に、ポアソン回帰の結果を使って種子数 y の予測線を描画し PNG ファイルへ出力してみます。
まず、種子数 y の予測値を算出するために、data3a.csv における x の最小値 7.19 から最大値 12.4 までを 0.1 刻みにした配列 xx を作成しました。
coef 関数を使えば glm の結果から Coefficients の Estimate 値を配列として取得できるので、これと xx を使って種子数 y の予測値を算出できます。
plot と layer を組み合わせる事で複数のグラフを重ねて描画できます。
poissonGlm_draw1.jl
using DataFrames, GLM, Gadfly d = readtable("data3a.csv") res = glm(y ~ x, d, Poisson(), LogLink()) # x の最小値 7.19 ~ 最大値 12.4 まで 0.1 刻みのデータを用意 xx = [minimum(d[:x]):0.1:maximum(d[:x])] # 種子数 y の予測値を算出 yy = exp(coef(res)[1] + coef(res)[2] * xx) # グラフの描画 p = plot( # data3a.csv の内容を描画 layer(d, x = "x", y = "y", color = "f", Geom.point), # 予測線の描画 layer(DataFrame(x = xx, y = yy), x = "x", y = "y", Geom.line) ) # PNG 形式で出力 draw(PNG("poissonGlm_draw1.png", 500px, 400px), p)

ここで、xx は以下のような内容です。
xx の内容
53-element Array{Float64,1}:
7.19
7.29
・・・
12.29
12.39
予測線の描画2
最後に、種子数 y の予測値を predict 関数で算出し、同様のグラフを描画します。
predict を使うには x の値だけでは無く (Intercept) の Estimate 値へ乗算する値も必要になるので、下記では DataFrame (下記の nd) へ項目 n (常に 1) として用意しました。
predict 内では のような計算が実施されます。
poissonGlm_draw2.jl
・・・ res = glm(y ~ x, d, Poisson(), LogLink()) # 7.19 ~ 12.4 まで 0.1 刻みのデータを用意 xx = [minimum(d[:x]):0.1:maximum(d[:x])] # predict へ渡すデータを作成 nd = DataFrame(n = [1 for i = 1:length(xx)], x = xx) # predict で種子数 y の予測値を算出 yy = predict(res, nd) p = plot( # data3a.csv の内容を描画 layer(d, x = "x", y = "y", color = "f", Geom.point), # 予測線の描画 layer(DataFrame(x = xx, y = yy), x = "x", y = "y", Geom.line) ) draw(PNG("poissonGlm_draw2.png", 500px, 400px), p)

Compiler Tree API で Java ソースファイルをパースする2 - Groovy で実装
前回 の処理を Groovy で実装してみました。
- Java SE 8u31
- Groovy 2.4.0
今回使用したソースは http://github.com/fits/try_samples/tree/master/blog/20150216/
(a) com.sun.tools.javac.main.JavaCompiler 利用
前回 Java で実装した内容を Groovy で実装し直しただけです。
javacompiler_parse.groovy
import javax.tools.ToolProvider import com.sun.tools.javac.main.JavaCompiler import com.sun.tools.javac.util.Context def fileManager = ToolProvider.systemJavaCompiler.getStandardFileManager(null, null, null) def compiler = new JavaCompiler(new Context()) fileManager.getJavaFileObjects(args).each { def cu = compiler.parse(it) cu.accept(new SampleVisitor(), null) }
groovy コマンドで実行するだけです。 当然ながら前回の Java 版と同じ結果になります。
実行結果
> groovy javacompiler_parse.groovy Sample.java
class: Sample
method: sample1
method: sample2
lambda: (n)->n * 2
class: SampleChild
method: child1
lambda: (n)->{
System.out.println(n);
return n > 0 && n % 2 == 0;
}
method: child2
(b) com.sun.source.util.JavacTask 利用
こちらも Groovy で実装し直しただけです。
javactask_parse.groovy
import javax.tools.ToolProvider def compiler = ToolProvider.systemJavaCompiler def fileManager = compiler.getStandardFileManager(null, null, null) def task = compiler.getTask(null, fileManager, null, null, null, fileManager.getJavaFileObjects(args)) task.parse().each { it.accept(new SampleVisitor(), null) }
ただし、こちらは groovy コマンドでスクリプト実行すると MissingMethodException が発生しました。
実行結果1
> groovy javactask_parse.groovy Sample.java Caught: groovy.lang.MissingMethodException: No signature of method: com.sun.tools.javac.tree.JCTree$JCCompilationUnit.accept() is applicable for argument types: (SampleVisitor, null) values: [SampleVisitor@2cf3d63b, null] Possible solutions: accept(com.sun.source.tree.TreeVisitor, java.lang.Object), accept(com.sun.tools.javac.tree.JCTree$Visitor), accept(com.sun.source.tree.TreeVisitor, java.lang.Object), accept(com.sun.tools.javac.tree.JCTree$Visitor), grep(), inspect() ・・・
JCCompilationUnit.accept(SampleVisitor, null) の箇所は javacompiler_parse.groovy でも同じだと思うのですが、こちらの方だけがエラーになる理由はよく分かりません。(Groovy の内部処理に何らかの原因があるのかもしれませんが)
ちなみに、groovyc でコンパイルして実行すると正常に実行できました。
実行結果2
> groovyc javactask_parse.groovy
> java -cp .;%JAVA_HOME%/lib/tools.jar;%GROOVY_HOME%/embeddable/groovy-all-2.4.0-indy.jar javactask_parse Sample.java
class: Sample
method: sample1
method: sample2
lambda: (n)->n * 2
class: SampleChild
method: child1
lambda: (n)->{
System.out.println(n);
return n > 0 && n % 2 == 0;
}
method: child2
Compiler Tree API で Java ソースファイルをパースする
javax.tools と Compiler Tree API を使って Java のソースファイルをパースしてみました。
- Java SE 8u31
前回 と同じように、ソースファイルを AST 化した CompilationUnitTree (実際は JCTree$JCCompilationUnit) を取得し、簡単な TreeVisitor を適用します。
今回のソースは http://github.com/fits/try_samples/tree/master/blog/20150208/
はじめに
ソースファイルを CompilationUnitTree 化する一般的な方法が分からなかったので、とりあえず以下のような方法で試してみました。 (com.sun.tools.javac.main.JavaCompiler は Compiler Tree API ではありませんが)
- (a) com.sun.tools.javac.main.JavaCompiler を利用
- (b) com.sun.source.util.JavacTask を利用
今回は、取得した CompilationUnitTree へ下記の TreeVisitor 実装クラスを適用します。
SampleVisitor.java
import com.sun.source.util.TreeScanner; import com.sun.source.tree.*; public class SampleVisitor extends TreeScanner<Void, Void> { @Override public Void visitClass(ClassTree node, Void p) { // クラス名の出力 System.out.println("class: " + node.getSimpleName()); return super.visitClass(node, p); } @Override public Void visitMethod(MethodTree node, Void p) { // メソッド名の出力 System.out.println("method: " + node.getName()); return super.visitMethod(node, p); } @Override public Void visitLambdaExpression(LambdaExpressionTree node, Void p) { // ラムダ式の内容を出力 System.out.println("lambda: " + node); return super.visitLambdaExpression(node, p); } }
SampleVisitor は以下の内容を出力します。
(a) com.sun.tools.javac.main.JavaCompiler 利用
com.sun.tools.javac.main.JavaCompiler を使った処理内容は以下の通りです。
- (1) javax.tools.ToolProvider から javax.tools.JavaCompiler 取得
- (2) javax.tools.JavaCompiler から StandardJavaFileManager 取得
- (3) StandardJavaFileManager から JavaFileObject (の Iterable) 取得
- (4) JavaFileObject を com.sun.tools.javac.main.JavaCompiler で parse して CompilationUnitTree (JCTree$JCCompilationUnit) を取得
- (5) CompilationUnitTree へ TreeVisitor を適用
javax.tools.JavaCompiler と com.sun.tools.javac.main.JavaCompiler は別物なのでご注意ください。
StandardJavaFileManager の getJavaFileObjects メソッドはソースファイル名を可変長引数の文字列で与えれば JavaFileObject (の Iterable) を取得できるので、コマンドライン引数の args をそのまま渡しています。
JavaCompilerParse.java
import java.io.IOException; import javax.tools.ToolProvider; import javax.tools.StandardJavaFileManager; import com.sun.source.tree.CompilationUnitTree; import com.sun.tools.javac.main.JavaCompiler; import com.sun.tools.javac.util.Context; public class JavaCompilerParse { public static void main(String... args) throws IOException { try ( // (1) (2) StandardJavaFileManager fileManager = ToolProvider.getSystemJavaCompiler() .getStandardFileManager(null, null, null) ) { JavaCompiler compiler = new JavaCompiler(new Context()); // (3) fileManager.getJavaFileObjects(args).forEach(f -> { // (4) AST 取得 CompilationUnitTree cu = compiler.parse(f); // (5) クラス・メソッド名とラムダ式の出力 cu.accept(new SampleVisitor(), null); }); } } }
JDK の lib/tools.jar を CLASSPATH へ指定してコンパイルする必要があります。
コンパイル
> javac -cp %JAVA_HOME%/lib/tools.jar *.java
実行する際も lib/tools.jar が必要です。
今回は適当に用意した Sample.java をパースしてみました。
実行結果
> java -cp .;%JAVA_HOME%/lib/tools.jar JavaCompilerParse Sample.java
class: Sample
method: sample1
method: sample2
lambda: (n)->n * 2
class: SampleChild
method: child1
lambda: (n)->{
System.out.println(n);
return n > 0 && n % 2 == 0;
}
method: child2
使用した Sample.java の内容は以下の通りです。
Sample.java
import java.util.stream.IntStream; class Sample { public String sample1() { return "sample"; } public int sample2(int x) { return IntStream.range(0, x).map(n -> n * 2).sum(); } class SampleChild { public void child1(int... nums) { IntStream.of(nums).filter(n -> { System.out.println(n); return n > 0 && n % 2 == 0; }).forEach(System.out::println); } private void child2() { } } }
(b) com.sun.source.util.JavacTask 利用
com.sun.source.util.JavacTask には、CompilationUnitTree (の Iterable) を取得する parse メソッドが用意されていますが、JavacTask を取得する一般的な方法が分からなかったので、今回は javax.tools.JavaCompiler の getTask メソッドで取得した JavaCompiler.CompilationTask の実装オブジェクト (com.sun.tools.javac.api.JavacTaskImpl) を JavacTask へキャストしました。
- (1) javax.tools.ToolProvider から javax.tools.JavaCompiler 取得
- (2) javax.tools.JavaCompiler から StandardJavaFileManager 取得
- (3) javax.tools.JavaCompiler を使って StandardJavaFileManager と JavaFileObject (の Iterable) から JavacTask 取得
- (4) JavacTask を parse して CompilationUnitTree (の Iterable) を取得
- (5) CompilationUnitTree へ TreeVisitor を適用
JavacTaskParse.java
import java.io.IOException; import javax.tools.ToolProvider; import javax.tools.JavaCompiler; import javax.tools.StandardJavaFileManager; import com.sun.source.util.JavacTask; public class JavacTaskParse { public static void main(String... args) throws IOException { // (1) JavaCompiler compiler = ToolProvider.getSystemJavaCompiler(); try ( // (2) StandardJavaFileManager fileManager = compiler.getStandardFileManager(null, null, null) ) { // (3) JavacTask task = (JavacTask)compiler.getTask(null, fileManager, null, null, null, fileManager.getJavaFileObjects(args)); // (4) (5) task.parse().forEach(cu -> cu.accept(new SampleVisitor(), null)); } } }
実行結果
> java -cp .;%JAVA_HOME%/lib/tools.jar JavacTaskParse Sample.java
class: Sample
method: sample1
method: sample2
lambda: (n)->n * 2
class: SampleChild
method: child1
lambda: (n)->{
System.out.println(n);
return n > 0 && n % 2 == 0;
}
method: child2