R で個体差のあるロジスティック回帰1 - glmmML
前回 のロジスティック回帰に続き、書籍 「 データ解析のための統計モデリング入門――一般化線形モデル・階層ベイズモデル・MCMC (確率と情報の科学) 」のサンプルを使って個体差を考慮したロジスティック回帰を GLMM と階層ベイズモデルで試してみます。
- (1) GLMM によるロジスティック回帰 (glmmML 関数)
- (2) MCMCglmm を使った階層ベイズモデルによるロジスティック回帰(MCMCglmm 関数)
ただし、今回は (1) だけで (2) に関しては次回に書く予定です。
サンプルソースは http://github.com/fits/try_samples/tree/master/blog/20140209/
はじめに
パッケージのインストール
今回使用するパッケージを R へインストールしておきます。
install.packages("glmmML")
データ
データは書籍のサポート web サイトから取得します。 今回は 7章「一般化線形混合モデル(GLMM)- 個体差のモデリング -」のサンプルデータを使います。
データ data7.csv
"N","y","x","id" 8,0,2,1 8,1,2,2 8,2,2,3 ・・・
データ内容は下記の通りです。
| 項目 | 内容 |
|---|---|
| N | 調査種子数 |
| y | 生存種子数 |
| x | 植物の葉数 |
| id | 個体のID |
葉数 の値が 2 ~ 6 までとなっており、
葉数毎に 20 個体ずつの生存種子数
をサンプリングしたデータとなっています。 (合計 100 個体)
二項分布の性質と過分散
ところで、二項分布の性質として下記があります。
今回のケースでは n が調査種子数(= 8)、p が生存確率です。
- (1) 平均(期待値とするのが適切かも)が
- (2) 分散が
ここで、今回のデータは下記のようになっています。
葉数 4(x = 4)場合の平均と分散
> mean(d[d$x == 4,]$y) [1] 4.05 > var(d[d$x == 4,]$y) [1] 8.365789
np = 4.05 とすると p = 4.05 / 8 = 約 0.5 となり、二項分布として期待される分散は np(1 - p) = 8 × 0.5 × (1 - 0.5) = 2 で、実際の分散 8.36 の方が明らかに大きい値となっています。 (過分散)
二項分布として考えると、原因不明な個体差等による効果を考慮した統計モデルを使ったロジスティック回帰が必要となり、GLMM や階層ベイズを使用するという流れとなるようです。
(1) GLMM によるロジスティック回帰 (glmmML 関数)
それでは glmmML() 関数を使った推定値の算出とグラフ化を実施してみます。
推定方法
個体差を考慮するため、GLMM では個体差のパラメータ を追加した線形予測子
を使う事になるようです。
ここで、 は生存確率です。
そして、 が正規分布の確率分布に従っていると仮定し、
確率密度関数 を加味して
を積分した
尤度 に対して
全データの積 の対数尤度
が最大になるようなパラメータ
の最尤推定値を求めます。
なお、 は二項分布の確率密度関数で、
は個体差
のばらつき(標準偏差)です。
glmmML() による推定
glmmML() 関数へ指定するオプションは、前回 の glm() とほとんど同じですが 、下記の点が異なります。
- 個体差のパラメータ
部分を cluster オプションで指定
今回は個体毎に異なる値が設定されている id 列を cluster オプションへ指定します。
logiGlmmML.R
d <- read.csv('data7.csv') library(glmmML) d.res <- glmmML(cbind(y, N - y) ~ x, data = d, family = binomial, cluster = id) summary(d.res) ・・・
glmmML() による推定結果は下記の通りです。
実行結果
Call: glmmML(formula = cbind(y, N - y) ~ x, family = binomial, data = d, cluster = id)
coef se(coef) z Pr(>|z|)
(Intercept) -4.190 0.8777 -4.774 1.81e-06
x 1.005 0.2075 4.843 1.28e-06
Scale parameter in mixing distribution: 2.408 gaussian
Std. Error: 0.2202
LR p-value for H_0: sigma = 0: 2.136e-55
Residual deviance: 269.4 on 97 degrees of freedom AIC: 275.4
と推定されました。
なお、glmmML() の結果から、
の値は
$coefficients で、 の値は
$sigma で取得できます。
葉数と生存種子数
推定したパラメータを使って葉数 x と生存種子数 y の予測線をグラフ化する処理は下記のようになります。
生存種子数の予測値は 調査種子数(= max(d$N) = 8)× 生存確率 で算出し、
生存確率 は
の式を元に算出します。
logiGlmmML.R
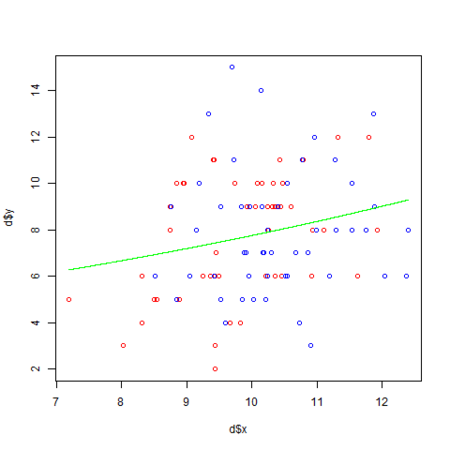
・・・ # 生存確率の算出 calcProb <- function(x, b, r) 1.0 / (1.0 + exp(-1 * (b[1] + b[2] * x + r))) png("logiGlmmML_1.png") # x と y をプロット plot(d$x, d$y) # x の範囲(2 ~ 6)を 100分割 xx <- seq(min(d$x), max(d$x), length = 100) beta <- d.res$coefficients # 個体差 r = 0 の葉数と生存種子数との予測線 lines(xx, max(d$N) * calcProb(xx, beta, 0), col="green") # 個体差 r = -2.408 の葉数と生存種子数との予測線 lines(xx, max(d$N) * calcProb(xx, beta, -1 * d.res$sigma), col="blue") # 個体差 r = 2.408 の葉数と生存種子数との予測線 lines(xx, max(d$N) * calcProb(xx, beta, d.res$sigma), col="blue") dev.off() ・・・
個体差 r = 0 (個体差なし)とした場合の予測線を緑色で、 r = ±2.408 の予測線を青色で描画しています。

葉数  の生存種子数分布
の生存種子数分布
次に、葉数 x が 4 の場合の生存種子数 y と個体数の分布と予測線をグラフ化する処理は下記のようになります。
glmmML() の結果から、x = 4 の場合の y の確率分布を算出し、x = 4 のサンプル数(= 20)を乗算して予測線を描画しています。
y の確率分布は、0 ~ 8 のそれぞれの値に対して で算出します。 (二項分布 × 正規分布 を個体差
で積分)
下記では sapply() と integrate() 関数を使って上記の算出処理を実装しています。
なお、lower と upper オプションを使って積分範囲を から
としています。
logiGlmmML.R
・・・ png("logiGlmmML_2.png") # 0 ~ 8 yy <- 0:max(d$N) # 生存種子数 y と個体数の分布 plot(yy, table(d[d$x == 4,]$y), xlab="y", ylab="num") # 葉数 x を固定した場合の生存種子数 y の確率分布を算出 calcL <- function(ylist, xfix, n, b, s) sapply(ylist, function(y) integrate( f = function(r) dbinom(y, n, calcProb(xfix, b, r)) * dnorm(r, 0, s), lower = s * -10, upper = s * 10 )$value ) # 予測線 lines(yy, calcL(yy, 4, max(d$N), beta, d.res$sigma) * length(d[d$x == 4,]$y), col="red", type="b") dev.off()

今回はここまで。 MCMCglmm を使った階層ベイズは次回。
Gradle で Jetty9 を使用
今のところ Gradle 標準の jetty プラグインでは Jetty6 しか使えないようなので、Servlet 3.0 を使いたいケースでは不便です。
この場合、build.gradle で Jetty9 の起動処理を自前で実装する手も考えられますが、Gradle Jetty Plugin for Eclipse Jetty (gradle-jetty-eclipse-plugin) を使った方が簡単だと思います。 (他にも gradle-jetty9-plugin というのもあります )
gradle-jetty-eclipse-plugin を使うと gradle jettyEclipseRun で Jetty 9.0.6 を起動できます。
gradle-jetty-eclipse-plugin の使い方
使用するには、build.gradle へ下記の設定を追加するだけです。
- gradle-jetty-eclipse-plugin を使用するための buildscript を定義
jettyEclipseを apply plugin する
gradle-jetty-eclipse-plugin を使用するための build.gradle 設定
apply plugin: 'jettyEclipse' buildscript { repositories { jcenter() maven { url 'http://dl.bintray.com/khoulaiz/gradle-plugins' } } dependencies { classpath (group: 'com.sahlbach.gradle', name: 'gradle-jetty-eclipse-plugin', version: '1.9.+') } }
なお、jcenter() の部分は Jetty9 のモジュールを取得できるリポジトリであればよいようなので mavenCentral() でも問題ないようです。
Jetty9 の実行
それでは @WebServlet アノテーションを使った単純な Servlet を実行してみます。
サンプルソースは http://github.com/fits/try_samples/tree/master/blog/20140203/ に配置しています。
まずは build.gradle です。
gradle-jetty-eclipse-plugin の利用設定を追加し、providedCompile で Servlet 3.0 の API を指定しました。
build.gradle
apply plugin: 'jettyEclipse' buildscript { repositories { jcenter() maven { url 'http://dl.bintray.com/khoulaiz/gradle-plugins' } } dependencies { classpath (group: 'com.sahlbach.gradle', name: 'gradle-jetty-eclipse-plugin', version: '1.9.+') } } repositories { mavenCentral() } dependencies { providedCompile 'javax.servlet:javax.servlet-api:3.0.1' }
Servlet は "sample" という文字を出力するだけの単純な処理を実装しました。
SampleServlet.java
package fits.sample; import java.io.IOException; import javax.servlet.ServletException; import javax.servlet.annotation.WebServlet; import javax.servlet.http.HttpServlet; import javax.servlet.http.HttpServletRequest; import javax.servlet.http.HttpServletResponse; @WebServlet(urlPatterns = {"/sample"}) public class SampleServlet extends HttpServlet { @Override protected void doGet(HttpServletRequest req, HttpServletResponse res) throws ServletException, IOException { res.getWriter().print("sample"); } }
それでは gradle jettyEclipseRun で実行してみます。
ちなみに、標準 jetty プラグインのような jettyRun と jettyRunWar のような区別はありません。
実行
> gradle jettyEclipseRun :compileJava :processResources UP-TO-DATE :classes :war :jettyEclipseRun Empty contextPath !RequestLog Hit <ENTER> to reload the webapp. Hit r + <ENTER> to rebuild and reload the webapp. Hit R + <ENTER> to rebuild the webapp without reload > Building 80% > :jettyEclipseRun > Running at http://localhost:8080/
これで Jetty9 が起動し、http://localhost:8080/sample へ接続すると "sample" という文字が返ってくるはずです。
Commons OGNL でマッピング・フィルタリング・畳み込み
以前、Java でマッピング・フィルタリング・畳み込みを試しましたが、 今回は Commons OGNL を使って OGNL 式によるマッピング・フィルタリング・畳み込みを Groovy で試してみました。
サンプルソースは http://github.com/fits/try_samples/tree/master/blog/20140118/
はじめに
Commons OGNL ではマッピングとフィルタリング用の式は用意されてますが、今のところ畳み込みは用意されていないようです。 (一応、畳み込みはラムダ式 :[ e ] で実装できました)
| 処理 | 機能名 | OGNL式 |
|---|---|---|
| マッピング | Projection | e1.{ e2 } |
| フィルタリング | Selection | e1.{? e2 } |
| 畳み込み | - | - |
Selection には、マッチした最初の要素を返す e1.{^ e2 } や最後の要素を返す e1.{$ e2 } のようなバリエーションも用意されています。
サンプルで使用するモデルクラス
今回のサンプルでは下記のようなモデルクラスを使いました。
Order.groovy
import groovy.transform.* @CompileStatic class Order { List<OrderLine> lines = [] } @CompileStatic @Immutable class OrderLine { String code BigDecimal price = 0 }
groovyc でコンパイルしておきます。
コンパイル
> groovyc Order.groovy
マッピング・フィルタリング
OGNL 式を使ったマッピング・フィルタリング処理を Groovy スクリプトで試してみます。
まず、@Grab を使って普通に Ognl.getValue(<OGNL式>, <オブジェクト>) を実行しようとすると下記のようなエラーが発生します。
エラー内容
java.lang.IllegalArgumentException: Javassist library is missing in classpath! Please add missed dependency!
at org.apache.commons.ognl.OgnlRuntime.getCompiler(OgnlRuntime.java:210) ・・・
Caused by: java.lang.ClassNotFoundException: Unable to resolve class: javassist.ClassPool
at org.apache.commons.ognl.OgnlRuntime.classForName(OgnlRuntime.java:665)
・・・
これは、デフォルトで使用される DefaultClassResolver が SystemClassLoader から javassist.ClassPool をロードしようとする事に起因するようで、Servlet 等の Java EE Web アプリケーションで実行する際にも同様のエラーが発生します。 (war ファイル内の JAR からロードするような場合)
エラー内容から CLASSPATH に javassist.jar が含まれていないように思ってしまいますが、そうではありません。
DefaultClassResolver に原因があるので、自前の ClassResolver を使って作成した Context を使うようにすればエラーを回避できます。
map_filter_sample.groovy
@GrabResolver('http://repository.apache.org/snapshots/') @Grab('org.apache.commons:commons-ognl:4.0-SNAPSHOT') import org.apache.commons.ognl.Ognl import org.apache.commons.ognl.ClassResolver def data = new Order() data.lines << new OrderLine('1', 100) data.lines << new OrderLine('2', 200) data.lines << new OrderLine('3', 300) try { // (1) DefaultClassResolver が原因でエラーが発生 println Ognl.getValue('lines.{? #this.code == "2" }', data) } catch (e) { println '(1) ' + e //e.printStackTrace() } println '-----------------------' // エラー回避のために自前の ClassResolver を使って Context 作成 def ctx = Ognl.createDefaultContext(null, { String className, Map<String, Object> context -> Class.forName(className) } as ClassResolver) // (2) マッピング println '(2) ' + Ognl.getValue('lines.{ #this.price > 100 }', ctx, data) // (3) フィルタリング println '(3) ' + Ognl.getValue('lines.{? #this.price > 100 }', ctx, data) // (4) マッチした最初の要素 println '(4) ' + Ognl.getValue('lines.{^ #this.price > 100 }', ctx, data) // (5) マッチした最後の要素 println '(5) ' + Ognl.getValue('lines.{$ #this.price > 100 }', ctx, data) // (6) OGNL式をコンパイルして使用 def exprNode = Ognl.compileExpression(ctx, null, 'lines.{? #this.code not in {"2", "4"} }') println '(6) ' + Ognl.getValue(exprNode, data)
実行結果は下記の通りです。
実行結果
> groovy map_filter_sample.groovy (1) java.lang.IllegalArgumentException: Javassist library is missing in classpath! Please add missed dependency! ----------------------- (2) [false, true, true] (3) [OrderLine(2, 200), OrderLine(3, 300)] (4) [OrderLine(2, 200)] (5) [OrderLine(3, 300)] (6) [OrderLine(1, 100), OrderLine(3, 300)]
畳み込み
OGNL のラムダ記法 :[ 処理 ] を使って畳み込みを試しに実装してみました。
OGNL のラムダは引数を一つしか取れないようなので(ラムダ内の #this にバインドされます)、リストを使ってラムダ #fold へ引数 {<処理>, <値>, <要素リスト>} を渡すようにしています。
更に、OrderLine の price を合計するための処理もラムダで定義して (:[ #this[0] + #this[1].price ])、#fold へ渡しています。
fold_sample.groovy
・・・ // 畳み込みの OGNL 式 def foldOgnl = ''' #fold = :[ #this[2].size() > 0 ? #fold({ #this[0], #this[0]({ #this[1], #this[2][0] }), #this[2].subList(1, #this[2].size()) }) : #this[1] ], #fold({ :[ #this[0] + #this[1].price ], 0, lines }) ''' // 0 + 100 + 200 + 300 println Ognl.getValue(foldOgnl, ctx, data)
実行結果
> groovy fold_sample.groovy 600
なお、合計を計算するだけなら以下のようにした方がシンプルです。
println Ognl.getValue('#v = 0, lines.{ #v = #v + #this.price }, #v', ctx, data)
並列実行時の注意点
最後に、並列処理で実行する際の注意点です。
問題確認のために、下記 3種類の処理を 50回並列でそれぞれ行ってみて OGNL の処理結果が正しかったかどうかを true・false で返すようにしてみます。
- (1) 文字列の OGNL 式で Context を再利用して getValue を実行
- (2) 文字列の OGNL 式で Context を都度作成して getValue を実行
- (3) compileExpression した結果で getValue を実行
parallel_sample.groovy
@GrabResolver('http://repository.apache.org/snapshots/') @Grab('org.apache.commons:commons-ognl:4.0-SNAPSHOT') import org.apache.commons.ognl.Ognl import org.apache.commons.ognl.ClassResolver import groovyx.gpars.* def createData = { i -> def data = new Order() data.lines << new OrderLine('1', i) data.lines << new OrderLine('2', i + 1) data.lines << new OrderLine('3', i + 2) data } def sum = { data -> data.lines.inject(0){ acc, val -> acc + val.price } } def printResult = { it.groupBy().each { k, v -> println "${k} ${v.size()}" } } def createContext = { Ognl.createDefaultContext(null, { String className, Map<String, Object> context -> Class.forName(className) } as ClassResolver) } def ctx = createContext() // OGNL式をコンパイル def exprNode = Ognl.compileExpression(ctx, null, '#v = 0, lines.{ #v = #v + #this.price }, #v') def count = 50 GParsPool.withPool(20) { // (1) 文字列の OGNL 式で並列処理(Context再利用) def res1 = (0..<count).collectParallel { def d = createData(it) try { // 稀に NoSuchPropertyException: Order.price が発生するため try-catch sum(d) == Ognl.getValue('#v = 0, lines.{ #v = #v + #this.price }, #v', ctx, d) } catch (e) { println e false } } // (2) 文字列の OGNL 式で並列処理(Context毎回作成) def res2 = (0..<count).collectParallel { def d = createData(it) sum(d) == Ognl.getValue('#v = 0, lines.{ #v = #v + #this.price }, #v', createContext(), d) } // (3) compileExpression 結果で並列処理 def res3 = (0..<count).collectParallel { def d = createData(it) sum(d) == Ognl.getValue(exprNode, d) } println '----- (1) 文字列の OGNL 式で並列処理(Context再利用)------' printResult res1 println '----- (2) 文字列の OGNL 式で並列処理(Context毎回作成) ---' printResult res2 println '----- (3) compileExpression 結果で並列処理 ----------------' printResult res3 }
処理結果は以下のようになり、(1) のケースで問題が発生しています。
実行結果
> groovy parallel_sample.groovy org.apache.commons.ognl.NoSuchPropertyException: Order.price ----- (1) 文字列の OGNL 式で並列処理(Context再利用)------ true 36 false 14 ----- (2) 文字列の OGNL 式で並列処理(Context毎回作成) --- true 50 ----- (3) compileExpression 結果で並列処理 ---------------- true 50
これは (1) のように Context を再利用すると #v や #this を並列処理間で共有してしまう事が原因と考えられます。
そのため、並列処理で実行する際は Context をその都度作成するか、compileExpression した結果を使う事になると思います。 (他の方法もあるかもしれません)
MathJax + AngularJS で数式の編集と表示
LaTeX の数式表示に JavaScript・HTML・CSS といった Web 技術で数式を表示できる MathJax が便利です。
簡易な数式編集ツールなら簡単に作れそうだったので、AngularJS を試すついでに以下のようなサンプルを作ってみました。
- TextArea に入力した数式を動的にプレビュー表示
サンプルソースは http://github.com/fits/try_samples/tree/master/blog/20140111/
数式編集のサンプル
MathJax で特定の要素の数式表示を更新するには下記のように処理します。
- (1) MathJax が適用された要素を取得
- (2)
MathJax.Hub.Queue()を使って (1) の要素へ数式を表示
なお、数式を表示する要素には MathJax の適用対象となるように ${}$ を設定しておきます。
下記サンプルでは、TextArea の内容に変化が生じるとプレビュー表示を更新するよう AngularJS の ng-change を使いました。
index.html
<!doctype html> <html ng-app> <head> <title>MathJax + AngularJS sample</title> <script type="text/x-mathjax-config"> MathJax.Hub.Config({ tex2jax: {inlineMath: [["$","$"],["\\(","\\)"]]} }); </script> <script src="http://cdn.mathjax.org/mathjax/latest/MathJax.js?config=TeX-AMS-MML_HTMLorMML"></script> <script src="https://ajax.googleapis.com/ajax/libs/angularjs/1.2.8/angular.min.js"></script> <script> function mathjax($scope) { $scope.formula = ''; $scope.changeFormula = function(formula) { // (1) 要素の取得 var el = MathJax.Hub.getAllJax('result')[0]; // (2) 数式の表示 MathJax.Hub.Queue(['Text', el, formula])(); }; } </script> </head> <body> <div ng-controller="mathjax"> <textarea ng-model="formula" ng-change="changeFormula(formula)"></textarea> </div> <br /> <!-- プレビュー表示 --> <div id="result">${}$</div> </body> </html>
画面例

R でロジスティック回帰 - glm, MCMCpack
前回 に続き、今回も書籍 「 データ解析のための統計モデリング入門――一般化線形モデル・階層ベイズモデル・MCMC (確率と情報の科学) 」のサンプルを使って GLM とベイズ統計を試してみます。
題材は、6章「GLM の応用範囲をひろげる -ロジスティック回帰など-」のサンプルデータを使ったロジスティック回帰です。
- (1) GLM によるロジスティック回帰 (glm 関数)
- (2) MCMCpack を使ったベイズ統計によるロジスティック回帰(MCMCmetrop1R 関数)
ここで、MCMCpack のロジスティック回帰に MCMClogit 関数を使えると思ったのですが、使い方がよく分からなかったので MCMCmetrop1R 関数のみ利用しました。
サンプルソースは http://github.com/fits/try_samples/tree/master/blog/20131229/
はじめに
MCMCpack パッケージを install.packages("MCMCpack") で R の実行環境へインストールしておきます。
ロジスティック回帰を試すためのデータは書籍のサポート web サイトから取得できます。
データ data4a.csv
N,y,x,f 8,1,9.76,C 8,6,10.48,C 8,5,10.83,C ・・・
データ内容は下記のようになっており、個体 i それぞれにおいて 「 個の観察種子のうち生きていて発芽能力があるものは
個、死んだ種子は
個」 となっています。
| 項目 | 内容 |
|---|---|
| N | 観察種子数 |
| y | 生存種子数 |
| x | 植物の体サイズ |
| f | 施肥処理 (C: 肥料なし, T: 肥料あり) |
体サイズ x と肥料による施肥処理 f が種子の生存する確率(ある個体 i から得られた 1個の種子が生存している確率)にどのように影響しているかをロジスティック回帰で解析します。
(1) GLM によるロジスティック回帰 (glm 関数)
まず、glm 関数を使ったロジスティック回帰を行って予測値の曲線を描画します。
特徴は以下のようになります。
- family に binomial (二項分布)を指定 (デフォルトのリンク関数が logit となる)
- family が binomial で応答変数 (
cbind(y, N - y)の箇所) が 2値(0, 1)以外なら cbind する必要あり (2値なら y だけでよい)
下記では、生存する確率 の関数が
、
線形予測子 で解析する事になります。
logisticGlm.R
d <- read.csv('data4a.csv') d.res <- glm(cbind(y, N - y) ~ x + f, data = d, family = binomial) summary(d.res) png("logisticGlm.png") # C: 肥料なしを 赤、T: 肥料ありを 青 で描画 plot(d$x, d$y, col = c("red", "blue")[d$f]) xx <- seq(min(d$x), max(d$x), length = 1000) ft <- factor("T", levels = levels(d$f)) fc <- factor("C", levels = levels(d$f)) # 下記でも可 #ft <- factor("T", levels = c("C", "T")) #fc <- factor("C", levels = c("C", "T")) # T: 肥料ありの予測値を算出 qq.t <- predict(d.res, data.frame(x = xx, f = ft), type="response") # C: 肥料なしの予測値を算出 qq.c <- predict(d.res, data.frame(x = xx, f = fc), type="response") # T: 肥料ありの予測値の曲線を 緑 で描画 lines(xx, max(d$N) * qq.t, col = "green") # C: 肥料なしの予測値の曲線を 黄 で描画 lines(xx, max(d$N) * qq.c, col = "yellow") dev.off()
predict 関数を使えば glm 結果の回帰モデルから新しいデータによる予測値を求めることができますので、"T:肥料あり" と "C:肥料なし" のそれぞれの予測値を求めて曲線を描画しています。
predict の結果は、生存する確率 の予測値ですので、縦軸が生存種子数
のグラフへ描画するには観察種子数 N (ここでは 8)を乗算する事になります。
ここで、"T:肥料あり" と "C:肥料なし" は factor 関数を使ってファクタ(因子)として作成しています。
d$f と同じ水準を持たせる必要がありますので levels 指定しています。
また、predict には glm で使った説明変数と同じ名前(上記の x と f)を使ったデータを渡す点に注意が必要です。
実行
実行すると下記のような結果になりました。
> R CMD BATCH logisticGlm.R
実行結果 logisticGlm.Rout
・・・
> d.res <- glm(cbind(y, N - y) ~ x + f, data = d, family = binomial)
>
> summary(d.res)
Call:
glm(formula = cbind(y, N - y) ~ x + f, family = binomial, data = d)
Deviance Residuals:
Min 1Q Median 3Q Max
-3.2584 -0.7948 0.1753 0.8757 3.1589
Coefficients:
Estimate Std. Error z value Pr(>|z|)
(Intercept) -19.5361 1.4138 -13.82 <2e-16 ***
x 1.9524 0.1389 14.06 <2e-16 ***
fT 2.0215 0.2313 8.74 <2e-16 ***
---
Signif. codes: 0 '***' 0.001 '**' 0.01 '*' 0.05 '.' 0.1 ' ' 1
(Dispersion parameter for binomial family taken to be 1)
Null deviance: 499.23 on 99 degrees of freedom
Residual deviance: 123.03 on 97 degrees of freedom
AIC: 272.21
Number of Fisher Scoring iterations: 5
・・・
この結果より、線形予測子 のパラメータ推定値
が
-19.5361, 1.9524, 2.0215 となります。

(2) MCMCpack を使ったベイズ統計によるロジスティック回帰(MCMCmetrop1R 関数)
次は、MCMCpack の MCMCmetrop1R 関数を使ったロジスティック回帰です。
今回のモデルの対数尤度は で、
、
なので、
これらを関数化 (下記の func) して MCMCmetrop1R 関数へ渡しています。
ちなみに、 です。(combination)
前回は、項目毎のデータ(d$x や d$y)を func 関数へ渡すようにしていましたが、今回はデータ d を丸ごと渡すようにしました。 MCMCmetrop1R(・・・, data = d, ・・・)
ファクタ(因子)のデータ (d$f) を直接計算に使えないようなので as.numeric で数値化して使っています。
なお、 の部分は
dbinom と log 関数を使うとシンプルになります。
パラメータ
の初期値は
theta.init で c(0, 0, 0) と指定しました。
logisticMcmcMetrop.R
library(MCMCpack) d <- read.csv('data4a.csv') func <- function(beta, data) { z <- beta[1] + beta[2] * data$x + beta[3] * as.numeric(data$f) q <- 1.0 / (1.0 + exp(-z)) sum(log(choose(data$N, data$y)) + data$y * log(q) + (data$N - data$y) * log(1 - q)) # 下記でも可 # sum(log(dbinom(data$y, data$N, q))) } d.res <- MCMCmetrop1R(func, theta.init = c(0, 0, 0), data = d, logfun = TRUE) summary(d.res) png("logisticMcmcMetrop.png") plot(d$x, d$y, col = c("red", "blue")[d$f]) xx <- seq(min(d$x), max(d$x), length = 1000) ft <- factor("T", levels = levels(d$f)) fc <- factor("C", levels = levels(d$f)) zt <- mean(d.res[,1]) + mean(d.res[,2]) * xx + mean(d.res[,3]) * as.numeric(ft) zc <- mean(d.res[,1]) + mean(d.res[,2]) * xx + mean(d.res[,3]) * as.numeric(fc) lines(xx, max(d$N) * 1.0 / (1.0 + exp(-zt)), col="green") lines(xx, max(d$N) * 1.0 / (1.0 + exp(-zc)), col="yellow") dev.off()
MCMCmetrop1R 結果の各パラメータの mean の値を使って、
肥料の有無(T と C)の生存確率の予測値をそれぞれ算出して、
観察種子数 N (ここでは 8)を乗算し曲線を描画しています。
実行
実行すると下記のような結果になります。
> R CMD BATCH logisticMcmcMetrop.R
実行結果 logisticMcmcMetrop.Rout
・・・
@@@@@@@@@@@@@@@@@@@@@@@@@@@@@@@@@@@@@@@@@@@@@@@@@@@@@@@@@
The Metropolis acceptance rate was 0.44590
@@@@@@@@@@@@@@@@@@@@@@@@@@@@@@@@@@@@@@@@@@@@@@@@@@@@@@@@@
>
> summary(d.res)
Iterations = 501:20500
Thinning interval = 1
Number of chains = 1
Sample size per chain = 20000
1. Empirical mean and standard deviation for each variable,
plus standard error of the mean:
Mean SD Naive SE Time-series SE
[1,] -21.694 1.5583 0.0110185 0.037186
[2,] 1.966 0.1386 0.0009798 0.003322
[3,] 2.027 0.2268 0.0016035 0.005383
2. Quantiles for each variable:
2.5% 25% 50% 75% 97.5%
var1 -24.836 -22.733 -21.667 -20.596 -18.766
var2 1.703 1.870 1.963 2.059 2.248
var3 1.594 1.873 2.022 2.172 2.494
・・・
Mean の値が glm の結果と似たような値になりました。 グラフにすると違いが分からないくらいです。

 の分布
の分布

 の分布
の分布

 の分布
の分布

R でポアソン回帰 - glm, MCMCpack
書籍 「 データ解析のための統計モデリング入門――一般化線形モデル・階層ベイズモデル・MCMC (確率と情報の科学) 」の 3章 「一般化線形モデル(GLM)」 と 9章 「GLMのベイズモデル化と事後分布の推定」 で説明されていたポアソン回帰を下記のような 3通りで試してみました。
- (1) GLM によるポアソン回帰 (glm 関数)
- (2) MCMCpack を使ったベイズ統計によるポアソン回帰1 (MCMCpoisson 関数)
- (3) MCMCpack を使ったベイズ統計によるポアソン回帰2 (MCMCmetrop1R 関数)
書籍では、R から WinBUGS を呼び出して MCMC サンプリングを行っていましたが、今回は R 上でベイズ統計解析を実施する MCMCpack パッケージを試してみました。
サンプルソースは http://github.com/fits/try_samples/tree/master/blog/20131215/
はじめに
まず、MCMCpack パッケージを使用するためにパッケージを R の実行環境へインストールしておきます。
MCMCpack のインストール
> install.packages("MCMCpack")
次に、ポアソン回帰を試すデータを書籍のサポート web サイトから取得してきます。
データ data3a.csv
y,x,f 6,8.31,C 6,9.44,C 6,9.5,C 12,9.07,C 10,10.16,C ・・・
ここで、データの内容は下記のようになっており、体サイズ x と肥料による施肥処理 f が種子数 y にどのように影響しているかをポアソン回帰で解析します。
| 項目 | 内容 |
|---|---|
| y | 種子数 |
| x | 植物の体サイズ |
| f | 施肥処理 |
(1) GLM によるポアソン回帰 (glm 関数)
それでは glm 関数を使ったポアソン回帰を試してみます。
ここでは stepAIC() を使った AIC によるモデル選択を試してみました。
poissonGlm.R
d <- read.csv('data3a.csv') # 説明変数を全て投入したモデル (y ~ x + f と同じ) d.all <- glm(y ~ ., data = d, family = poisson) library(MASS) # AIC によるモデル選択 d.res <- stepAIC(d.all) summary(d.res) png("poissonGlm.png") plot(d$x, d$y, col = c("red", "blue")[d$f]) xx <- seq(min(d$x), max(d$x), length = 1000) # y ~ x のモデルを使った平均種子数の予測値の曲線を描画 lines(xx, exp(d.res$coefficients["(Intercept)"] + d.res$coefficients["x"] * xx), col="green") # 以下でも可 #lines(xx, predict(d.res, newdata = data.frame(x = xx), type = "response"), col = "green") dev.off()
なお、今回は y ~ x のモデルが選択される事 (施肥処理は効果が無い) を予め分かっているので、y ~ x のモデルを使って平均種子数の予測値を描画 (緑の線) しています。
ここで、 y ~ x モデルのリンク関数 (対数リンク関数) は で、平均種子数
は
となります。
また、 の最尤推定値が
d.res$coefficients["(Intercept)"]、 の最尤推定値が
d.res$coefficients["x"] に該当します。
実行
実行すると下記のような結果になります。
> R CMD BATCH poissonGlm.R
実行結果 poissonGlm.Rout
・・・
> d.res <- stepAIC(d.all)
Start: AIC=476.59
y ~ x + f
Df Deviance AIC
- f 1 84.993 474.77
<none> 84.808 476.59
- x 1 89.475 479.25
Step: AIC=474.77
y ~ x
Df Deviance AIC
<none> 84.993 474.77
- x 1 89.507 477.29
>
> summary(d.res)
Call:
glm(formula = y ~ x, family = poisson, data = d)
Deviance Residuals:
Min 1Q Median 3Q Max
-2.3679 -0.7348 -0.1775 0.6987 2.3760
Coefficients:
Estimate Std. Error z value Pr(>|z|)
(Intercept) 1.29172 0.36369 3.552 0.000383 ***
x 0.07566 0.03560 2.125 0.033580 *
---
Signif. codes: 0 '***' 0.001 '**' 0.01 '*' 0.05 '.' 0.1 ' ' 1
(Dispersion parameter for poisson family taken to be 1)
Null deviance: 89.507 on 99 degrees of freedom
Residual deviance: 84.993 on 98 degrees of freedom
AIC: 474.77
Number of Fisher Scoring iterations: 4
・・・
この結果より、平均種子数の予測値を求める関数は になります。

(2) MCMCpack を使ったベイズ統計によるポアソン回帰1 (MCMCpoisson 関数)
MCMCpack でポアソン回帰を行うには MCMCpoisson 関数を使うのが簡単だと思います。 基本的に glm 関数と同様のモデル式とデータを指定するだけです。
ここでは y ~ x のモデルだけをポアソン回帰してみました。
なお、glm 関数では線形予測子のパラメータ と
の最尤推定値を取得しますが、MCMCpoisson 関数では
と
のそれぞれの分布を取得する点が大きく異なります。
poissonMcmcPoisson.R
library(MCMCpack) d <- read.csv('data3a.csv') d.res <- MCMCpoisson(y ~ x, data = d) summary(d.res) png("poissonMcmcPoisson.png") plot(d$x, d$y, col = c("red", "blue")[d$f]) xx <- seq(min(d$x), max(d$x), length = 10000) lines(xx, exp(mean(d.res[,1]) + mean(d.res[,2]) * xx), col="green") dev.off()
glm の時とは異なり、 と
のそれぞれの算術平均値 (
mean の結果) を使って、平均種子数の予測値を描画 (緑の線) しています。
実行
実行すると下記のような結果になります。
> R CMD BATCH poissonMcmcPoisson.R
実行結果 poissonMcmcPoisson.Rout
・・・
> summary(d.res)
Iterations = 1001:11000
Thinning interval = 1
Number of chains = 1
Sample size per chain = 10000
1. Empirical mean and standard deviation for each variable,
plus standard error of the mean:
Mean SD Naive SE Time-series SE
(Intercept) 1.29315 0.3602 0.003602 0.010757
x 0.07547 0.0352 0.000352 0.001047
2. Quantiles for each variable:
2.5% 25% 50% 75% 97.5%
(Intercept) 0.586606 1.05208 1.2956 1.54153 1.9917
x 0.006025 0.05125 0.0756 0.09951 0.1438
・・・
Mean の値が glm の結果とほぼ同じ値になっています。

ちなみに、d.res には MCMC サンプリング結果として 1万個の と
の値が格納されています。
 の分布
の分布

 の分布
の分布

(3) MCMCpack を使ったベイズ統計によるポアソン回帰2 (MCMCmetrop1R 関数)
最後に MCMCmetrop1R 関数を使ってみます。
MCMCmetrop1R 関数では自前で定義した尤度関数を使って MCMC サンプリングを実施できるので汎用的に使えます。
今回のモデルの対数尤度は で、
なので、これらを関数化 (下記の
func) しています。
尤度関数が対数尤度か否かは logfun で指定するようになっており (TRUE の場合が対数尤度)、デフォルト値は TRUE となっています。 (今回は対数尤度なので logfun = TRUE です)
また、theta.init で と
の初期値を
c(0, 0) と指定しています。
poissonMcmcMetrop.R
library(MCMCpack) d <- read.csv('data3a.csv') # 尤度関数(対数尤度関数) func <- function(beta, x, y) { lambda <- exp(beta[1] + beta[2] * x) sum(log(dpois(y, lambda))) } d.res <- MCMCmetrop1R(func, theta.init = c(0, 0), x = d$x, y = d$y, logfun = TRUE) # 下記でも同じ #d.res <- MCMCmetrop1R(func, theta.init = c(0, 0), x = d$x, y = d$y) summary(d.res) png("poissonMcmcMetrop.png") plot(d$x, d$y, col = c("red", "blue")[d$f]) xx <- seq(min(d$x), max(d$x), length = 10000) lines(xx, exp(mean(d.res[,1]) + mean(d.res[,2]) * xx), col="green") dev.off()
実行
実行すると下記のような結果になります。
> R CMD BATCH poissonMcmcMetrop.R
実行結果 poissonMcmcMetrop.Rout
・・・
> summary(d.res)
Iterations = 501:20500
Thinning interval = 1
Number of chains = 1
Sample size per chain = 20000
1. Empirical mean and standard deviation for each variable,
plus standard error of the mean:
Mean SD Naive SE Time-series SE
[1,] 1.29545 0.35797 0.0025313 0.0077477
[2,] 0.07528 0.03498 0.0002473 0.0007561
2. Quantiles for each variable:
2.5% 25% 50% 75% 97.5%
var1 0.584646 1.05368 1.29884 1.53850 1.9830
var2 0.007798 0.05159 0.07518 0.09907 0.1448
・・・
glm や MCMCpoisson と似たような結果となりました。
なお、MCMCpoisson と MCMCmetrop1R ではバーンイン burnin とサンプリング数 mcmc のデフォルト値が異なっています。
| 関数 | burnin のデフォルト値 | mcmc のデフォルト値 |
|---|---|---|
| MCMCpoisson | 1000 | 10000 |
| MCMCmetrop1R | 500 | 20000 |
Groovy で Dempsy を分散実行
前回 と 前々回に続き、今回は Dempsy を Groovy で分散実行してみます。
サンプルソースは http://github.com/fits/try_samples/tree/master/blog/20131208/
はじめに
Dempsy を分散実行するには ZooKeeper が必要となりますので 「GroovyでZooKeeperを組み込み実行」 で作成した Groovy スクリプトを使う事にします。
また、分散実行するには Dempsy オブジェクトの構成を下記のようにします。 (※ の箇所が単独実行と異なる部分)
Dempsy 分散実行時の構成
| プロパティ名 | クラス |
|---|---|
| clusterSessionFactory | ZookeeperSessionFactory ※ |
| clusterCheck | SpecificClusterCheck ※ |
| defaultRoutingStrategy | DecentralizedRoutingStrategy |
| defaultSerializer | KryoSerializer |
| defaultStatsCollectorFactory | StatsCollectorFactoryCoda |
| defaultTransport | TcpTransport ※ |
Dempsy 分散実行アプリケーション
それでは、前回の KeySource 指定無しのサンプルを元に分散実行への対応を行ってみます。
基本的には Dempsy オブジェクトの構成を変えるだけです。
単独実行と異なり、分散実行では ClusterDefinition 毎にプロセスを起動しますので、実行時引数でクラスター名を指定するようにしています。
なお、dempsy.waitToBeStopped() はクラスター adaptor が終了しないようにするための措置です。 (dempsy.waitToBeStopped() が無いと、すぐに終了してしまいます)
money_count_vertx_cluster.groovy
・・・前回と同じ実装・・・ // 実行する ClusterDefinition の名称 def cluster = args[0] def dempsy = new Dempsy() dempsy.applicationDefinitions = [app] dempsy.clusterSessionFactory = new ZookeeperSessionFactory('localhost:2181', 5000) // 実行する ClusterDefinition を選定 dempsy.clusterCheck = new SpecificClusterCheck(new ClusterId('money-count', cluster)) // mp を 3ノード構成で処理する設定 dempsy.defaultRoutingStrategy = new DecentralizedRoutingStrategy(5, 3) dempsy.defaultSerializer = new KryoSerializer() dempsy.defaultStatsCollectorFactory = new StatsCollectorFactoryCoda() dempsy.defaultTransport = new TcpTransport() dempsy.start() Runtime.runtime.addShutdownHook { -> println 'shutdown ...' dempsy.stop() } // Adaptor が終了するのを防止(MessageProcessor の場合は不要) dempsy.waitToBeStopped()
MessageProcessor (クラスター mp) の実行ノード数などは DecentralizedRoutingStrategy で設定します。
DecentralizedRoutingStrategy のコンストラクタは DecentralizedRoutingStrategy(int defaultTotalSlots, int defaultNumNodes) となっており、第1引数でスロット(シャード)数、第2引数でノード数を指定するようになっています。
キー毎の担当スロットが messageKey.hashCode() % defaultTotalSlots で算出され、ノード毎にスロットが割り当てられて処理されます。
今回のサンプルのキー構成でスロットが適切にバラけるように defaultTotalSlots を 5 にしました。
スロット数 5 の場合のキー毎の messageKey.hashCode() % 5 結果
| キー名 | スロット |
|---|---|
| 1 | 4 |
| 5 | 3 |
| 10 | 2 |
| 50 | 1 |
| 100 | 0 |
| 500 | 4 |
| 1000 | 3 |
| 2000 | 4 |
| 5000 | 2 |
| 10000 | 1 |
分散実行
それでは分散実行してみます。
まずは ZooKeeper を実行しておきます。
ZooKeeper 実行
> groovy zk_run.groovy zoo.cfg
次に Adaptor を実行します。
Adaptor 実行
> groovy money_count_vertx_cluster.groovy adaptor ・・・ MoneyAdaptor.start ... ・・・
そして MessageProcessor を実行します。
DecentralizedRoutingStrategy で指定したのは 3ノードですが、ノードが停止した場合の挙動を確認するため今回は 4ノード分のプロセスを実行しました。
(1) mp 実行 (1つ目)
> groovy money_count_vertx_cluster.groovy mp ・・・ 2013-12-08 19:42:11,187 [myid:] - INFO [main-SendThread(localhost:2181):ClientCnxn$SendThread@738] - Session establishment complete on server localhost/127.0.0.1:2181, sessionid = 0x142d19fda340012, negotiated timeout = 6000 2013-12-08 19:42:15,664 [myid:] - INFO [main:SimpleThreadPool@270] - Job execution threads will use class loader of thread: main ・・・
(2) mp 実行 (2つ目)
> groovy money_count_vertx_cluster.groovy mp ・・・
(3) mp 実行 (3つ目)
> groovy money_count_vertx_cluster.groovy mp ・・・
(4) mp 実行 (4つ目)
> groovy money_count_vertx_cluster.groovy mp ・・・
結果1 - 全キーへのアクセス
下記のように全キーの URL へアクセスしてカウントアップしてみます。
$ curl http://localhost:8080/1 $ curl http://localhost:8080/10 $ curl http://localhost:8080/100 $ curl http://localhost:8080/1000 $ curl http://localhost:8080/10000 $ curl http://localhost:8080/5000 $ curl http://localhost:8080/500 $ curl http://localhost:8080/50 $ curl http://localhost:8080/5 $ curl http://localhost:8080/2000
(1) ~ (4) の mp の状態と割り当てられたスロットは下記のようになりました。
なお、(4) のノードにはスロットが割り当てられておらずキーも送信されていない事を確認できました。
(1) mp の状態 (スロット 3)
> groovy money_count_vertx_cluster.groovy mp ・・・ MoneyCount.activation : 1000 key: 1000, count: 1 MoneyCount.activation : 5 key: 1000, count: 1 key: 5, count: 1 ・・・
(2) mp の状態 (スロット 0, 1)
> groovy money_count_vertx_cluster.groovy mp ・・・ MoneyCount.activation : 100 MoneyCount.activation : 10000 key: 100, count: 1 key: 10000, count: 1 MoneyCount.activation : 50 key: 100, count: 1 key: 10000, count: 1 key: 50, count: 1 ・・・
(3) mp の状態 (スロット 2, 4)
> groovy money_count_vertx_cluster.groovy mp ・・・ MoneyCount.activation : 1 MoneyCount.activation : 10 key: 1, count: 1 key: 10, count: 1 MoneyCount.activation : 5000 MoneyCount.activation : 500 key: 1, count: 1 key: 500, count: 1 key: 5000, count: 1 key: 10, count: 1 MoneyCount.activation : 2000 key: 1, count: 1 key: 500, count: 1 key: 2000, count: 1 key: 5000, count: 1 key: 10, count: 1 ・・・
(4) mp の状態 (変化なし)
> groovy money_count_vertx_cluster.groovy mp ・・・
結果2 - (1) の mp を停止
(1) の mp を Ctrl + c でプロセス終了して、下記 URL へアクセスしてみます。
$ curl http://localhost:8080/1000 $ curl http://localhost:8080/5
(1) の担当スロットが、代わりに (4) へ割り当てられてキーが送信されてくるようになります。
(4) mp の状態 ((1) の停止後)
> groovy money_count_vertx_cluster.groovy mp ・・・ MoneyCount.activation : 1000 MoneyCount.activation : 5 key: 1000, count: 1 key: 5, count: 1 ・・・
