Javaの列挙型(Enum)へ新しい要素を追加
Java の列挙型(Enum)へ新しい要素(識別子)を動的に追加する方法を探ってみました。
列挙型の場合、普通のリフレクションクラスではインスタンス化できませんので、下記のように sun パッケージのクラスを使用する必要があります。
- (1) sun.reflect.ConstructorAccessor で列挙型の新しい要素をインスタンス化
- (2) sun.misc.Unsafe で列挙型の $VALUES フィールドへ (1) のインスタンスを追加
そのため、下記の環境では動作確認できましたが、他の Java 実行環境では使えないかもしれません。
ソースは http://github.com/fits/try_samples/tree/master/blog/20140309/
はじめに
今回は下記のような列挙型へ Second と Third の要素を追加する事にします。
enum EType {
First
}
Constructor の newInstance では列挙型をインスタンス化できない
下記のように Constructor の newInstance メソッドで列挙型(今回の EType)の新しい要素をインスタンス化したいところなのですが、IllegalArgumentException エラーとなります。
EnumAddValueError.java
import java.lang.reflect.Constructor; public class EnumAddValueError { public static void main(String... args) throws Exception { // EType のコンストラクタ(private)取得 Constructor<EType> cls = EType.class.getDeclaredConstructor(String.class, int.class); // private コンストラクタを実行するための設定 cls.setAccessible(true); // 列挙型は newInstance できないのでエラー EType t2 = cls.newInstance("Second", 1); } enum EType { First } }
実行例
> java EnumAddValueError
Exception in thread "main" java.lang.IllegalArgumentException: Cannot reflectively create enum objects
at java.lang.reflect.Constructor.newInstance(Constructor.java:521)
at EnumAddValueError.main(EnumAddValueError.java:11)
これは newInstance メソッドの実装内容が原因です。
java.lang.reflect.Constructor のソース(一部抜粋)
public final class Constructor<T> extends Executable { ・・・ @CallerSensitive public T newInstance(Object ... initargs) throws InstantiationException, IllegalAccessException, IllegalArgumentException, InvocationTargetException { ・・・ if ((clazz.getModifiers() & Modifier.ENUM) != 0) throw new IllegalArgumentException("Cannot reflectively create enum objects"); ConstructorAccessor ca = constructorAccessor; // read volatile if (ca == null) { ca = acquireConstructorAccessor(); } @SuppressWarnings("unchecked") T inst = (T) ca.newInstance(initargs); return inst; } ・・・ }
ここで、sun.reflect.ConstructorAccessor を直接使えば IllegalArgumentException を避けてインスタンス化できる事も分かります。
実装
それでは、列挙型へ新しい要素を追加する処理を実装してみます。
(1) sun.reflect.ConstructorAccessor で列挙型の新しい要素をインスタンス化
まずは、列挙型(下記の EType)の新しいインスタンスを作成する必要がありますが、前述したように sun.reflect.ConstructorAccessor を使う必要があります。
ここで、どのようにして列挙型の ConstructorAccessor を入手するかが課題となりますが、今回は Constructor の acquireConstructorAccessor メソッドを使って取得してみました。
acquireConstructorAccessor は private メソッドなのでリフレクションを使って実行します。
列挙型の ConstructorAccessor が手に入れば、newInstance メソッドを実行して列挙型の新しいインスタンスを得る事ができます。
EnumAddValue.java (列挙型のインスタンス化)
・・・ import sun.reflect.ConstructorAccessor; public class EnumAddValue { public static void main(String... args) throws Exception { EType t2 = addEnumValue(EType.class, "Second", 1); ・・・ } // (1) sun.reflect.ConstructorAccessor で列挙型の新しい要素をインスタンス化 private static <T extends Enum<?>> T addEnumValue(Class<T> enumClass, String name, int ordinal) throws Exception { // acquireConstructorAccessor メソッド Method m = Constructor.class.getDeclaredMethod("acquireConstructorAccessor"); m.setAccessible(true); // 列挙型のコンストラクタ取得 Constructor<T> cls = enumClass.getDeclaredConstructor(String.class, int.class); // acquireConstructorAccessor を実行し ConstructorAccessor を取得 ConstructorAccessor ca = (ConstructorAccessor)m.invoke(cls); // 列挙型の新しい要素をインスタンス化 @SuppressWarnings("unchecked") T result = (T)ca.newInstance(new Object[]{name, ordinal}); // (2) sun.misc.Unsafe で列挙型の $VALUES フィールドへ (1) のインスタンスを追加 addValueToEnum(result); return result; } ・・・ enum EType { First } }
(2) sun.misc.Unsafe で列挙型の $VALUES フィールドへ (1) のインスタンスを追加
(1) で列挙型の新しい要素をインスタンス化できるようになりましたが、これだけでは不十分です。
valueOf メソッド等を使えるようにするには、列挙型の $VALUES クラスフィールド(private static final)へ新しいインスタンスを追加する必要があります。
private final なクラスフィールドの内容を強引に変更するには sun.misc.Unsafe クラスを使用する事になります。
ここで、Unsafe のインスタンスを Unsafe.getUnsafe() で取得したいところですが、今回のやり方だと SecurityException エラーとなってしまいます。
SecurityException を回避するのは面倒そうだったので、今回はリフレクションを使って Unsafe の theUnsafe クラスフィールド(private static final)から Unsafe インスタンスを取得してみました。
Unsafe のインスタンスを得られれば putObjectVolatile メソッド等で private final なクラスフィールドの内容を変更できます。
EnumAddValue.java ($VALUES への要素追加)
・・・
// (2) sun.misc.Unsafe で列挙型の $VALUES フィールドへ (1) のインスタンスを追加
private static <T extends Enum<?>> void addValueToEnum(T newValue) throws Exception {
// $VALUES フィールド取得
Field f = newValue.getClass().getDeclaredField("$VALUES");
f.setAccessible(true);
// $VALUES の値を取得
@SuppressWarnings("unchecked")
T[] values = (T[])f.get(null);
T[] newValues = Arrays.copyOf(values, values.length + 1);
// 列挙型の新しい要素を追加
newValues[values.length] = newValue;
// theUnsafe フィールド
Field uf = Unsafe.class.getDeclaredField("theUnsafe");
uf.setAccessible(true);
// theUnsafe フィールドから Unsafe インスタンスを取得
Unsafe unsafe = (Unsafe)uf.get(null);
// $VALUES フィールドへ値を設定
unsafe.putObjectVolatile(unsafe.staticFieldBase(f), unsafe.staticFieldOffset(f), newValues);
}
・・・
実行
今回作成したソースの全容は下記の通りです。
EnumAddValue.java
import java.lang.reflect.Constructor; import java.lang.reflect.Field; import java.lang.reflect.Method; import java.util.Arrays; import sun.misc.Unsafe; import sun.reflect.ConstructorAccessor; public class EnumAddValue { public static void main(String... args) throws Exception { EType t2 = addEnumValue(EType.class, "Second", 1); System.out.println(t2); EType t3 = addEnumValue(EType.class, "Third", 2); System.out.println(EType.valueOf("Third")); System.out.println("Thrid == t3 : " + (EType.valueOf("Third") == t3)); System.out.println("-----"); for (EType type : EType.values()) { System.out.println(type); } } // (1) sun.reflect.ConstructorAccessor で列挙型の新しい要素をインスタンス化 private static <T extends Enum<?>> T addEnumValue(Class<T> enumClass, String name, int ordinal) throws Exception { Method m = Constructor.class.getDeclaredMethod("acquireConstructorAccessor"); m.setAccessible(true); Constructor<T> cls = enumClass.getDeclaredConstructor(String.class, int.class); ConstructorAccessor ca = (ConstructorAccessor)m.invoke(cls); @SuppressWarnings("unchecked") T result = (T)ca.newInstance(new Object[]{name, ordinal}); addValueToEnum(result); return result; } // (2) sun.misc.Unsafe で列挙型の $VALUES フィールドへ (1) のインスタンスを追加 private static <T extends Enum<?>> void addValueToEnum(T newValue) throws Exception { Field f = newValue.getClass().getDeclaredField("$VALUES"); f.setAccessible(true); @SuppressWarnings("unchecked") T[] values = (T[])f.get(null); T[] newValues = Arrays.copyOf(values, values.length + 1); newValues[values.length] = newValue; Field uf = Unsafe.class.getDeclaredField("theUnsafe"); uf.setAccessible(true); Unsafe unsafe = (Unsafe)uf.get(null); unsafe.putObjectVolatile(unsafe.staticFieldBase(f), unsafe.staticFieldOffset(f), newValues); } enum EType { First } }
ビルドすると ConstructorAccessor・Unsafe を使っている事に対する警告が出ます。
ビルド
> javac EnumAddValue.java EnumAddValue.java:7: 警告: Unsafeは内部所有のAPIであり、今後のリリースで削除される可能性があります import sun.misc.Unsafe; ・・・ 警告7個
実行してみると、一応 Second と Third を追加できている事を確認できました。
実行
> java EnumAddValue Second Third Thrid == t3 : true ----- First Second Third
R でロジスティック回帰とオッズ比の算出 - glm, MCMClogit
以前、glm・MCMCmetrop1R 関数でロジスティック回帰を試みましたが、今回はその時に利用を断念した MCMCpack の MCMClogit 関数を使ってロジスティック回帰を行います。
題材は、書籍 「 データサイエンティスト養成読本 [ビッグデータ時代のビジネスを支えるデータ分析力が身につく! ] (Software Design plus) 」の p.38 と同様の Titanic データセットを使ったロジスティック回帰とオッズ比の算出です。
以前使ったデータは主に数値でしたが、今回は主に因子(factor)データを扱っている点が異なっています。
今回のソースは http://github.com/fits/try_samples/tree/master/blog/20140302/
はじめに
MCMCpack パッケージを R へインストールしておきます。
install.packages("MCMCpack")
Titanic データセット
Titanic のデータセットは R にデフォルトで用意されており、data.frame(Titanic) すると下記のようになります。
data.frame(Titanic) 結果
> data.frame(Titanic) Class Sex Age Survived Freq 1 1st Male Child No 0 2 2nd Male Child No 0 3 3rd Male Child No 35 4 Crew Male Child No 0 5 1st Female Child No 0 6 2nd Female Child No 0 7 3rd Female Child No 17 8 Crew Female Child No 0 9 1st Male Adult No 118 10 2nd Male Adult No 154 11 3rd Male Adult No 387 12 Crew Male Adult No 670 13 1st Female Adult No 4 14 2nd Female Adult No 13 15 3rd Female Adult No 89 16 Crew Female Adult No 3 17 1st Male Child Yes 5 18 2nd Male Child Yes 11 19 3rd Male Child Yes 13 20 Crew Male Child Yes 0 ・・・
内容は以下の通りです。
| Class(船室等級) | Sex(性別) | Age(年齢層) | Survived(生存可否) | Freq(人数) |
|---|---|---|---|---|
| 1st, 2nd, 3rd, Crew | Male, Female | Child, Adult | No, Yes | 数値 |
今回は、ロジスティック回帰の結果から船室等級・性別・年齢層毎の生存率に対するオッズ比を算出します。
(1) glm を使ったロジスティック回帰とオッズ比
書籍の内容とほとんど同じですが、
まずは glm 関数を使ったロジスティック回帰です。
Survived~. としているので Class・Sex・Age を説明変数としたロジスティック回帰を実施します。
オッズ比は exp(<推定値>) で算出できるので、書籍のような epicalc パッケージは使わず、glm 結果の $coefficients を exp 関数へ渡してオッズ比を算出しました。
logiMcmcglmm.R
d <- data.frame(Titanic) d.data <- data.frame( Class = rep(d$Class, d$Freq), Sex = rep(d$Sex, d$Freq), Age = rep(d$Age, d$Freq), Survived = rep(d$Survived, d$Freq) ) d.res <- glm(Survived~., data = d.data, family = binomial) summary(d.res) # オッズ比 exp(d.res$coefficients)
なお、上記では data.frame(Titanic) の Freq(人数) を展開したデータフレーム(下記)を使ってロジスティック回帰を実施しています。
d.data の内容
Class Sex Age Survived 1 3rd Male Child No 2 3rd Male Child No ・・・ 35 3rd Male Child No 36 3rd Female Child No ・・・
実行結果は下記の通りです。
実行結果
・・・
> d.res <- glm(Survived~., data = d.data, family = binomial)
> summary(d.res)
Call:
glm(formula = Survived ~ ., family = binomial, data = d.data)
Deviance Residuals:
Min 1Q Median 3Q Max
-2.0812 -0.7149 -0.6656 0.6858 2.1278
Coefficients:
Estimate Std. Error z value Pr(>|z|)
(Intercept) 0.6853 0.2730 2.510 0.0121 *
Class2nd -1.0181 0.1960 -5.194 2.05e-07 ***
Class3rd -1.7778 0.1716 -10.362 < 2e-16 ***
ClassCrew -0.8577 0.1573 -5.451 5.00e-08 ***
SexFemale 2.4201 0.1404 17.236 < 2e-16 ***
AgeAdult -1.0615 0.2440 -4.350 1.36e-05 ***
---
Signif. codes: 0 '***' 0.001 '**' 0.01 '*' 0.05 '.' 0.1 ' ' 1
(Dispersion parameter for binomial family taken to be 1)
Null deviance: 2769.5 on 2200 degrees of freedom
Residual deviance: 2210.1 on 2195 degrees of freedom
AIC: 2222.1
Number of Fisher Scoring iterations: 4
>
> # オッズ比
> exp(d.res$coefficients)
(Intercept) Class2nd Class3rd ClassCrew SexFemale AgeAdult
1.9844057 0.3612825 0.1690159 0.4241466 11.2465380 0.3459219
オッズ比から、女性(Female)は男性(Male)の 11倍(11.2465380)、大人(Adult)は子供(Child)の 3分の1 (0.3459219)の生存率という結果になりました。
(2) MCMClogit を使ったロジスティック回帰とオッズ比
それでは、MCMCpack の MCMClogit 関数を使って同様の処理を実施してみます。
glm 関数の場合とほとんど同じですが、MCMClogit 関数は応答変数(下記の Survived)に因子型(factor)を扱えないようなので、Survived の No と Yes を 0(= No) と 1(= Yes)へ変換しています。
(as.numeric(d$Survived) - 1 の箇所)
また、MCMClogit 関数の結果も glm とは違って値の分布となるので summary した結果の平均値(期待値) Mean を使ってオッズ比を算出しています。
logistic_odds_mcmclogit.R
library(MCMCpack) d <- data.frame(Titanic) d.data <- data.frame( Class = rep(d$Class, d$Freq), Sex = rep(d$Sex, d$Freq), Age = rep(d$Age, d$Freq), Survived = rep(as.numeric(d$Survived) - 1, d$Freq) ) d.res <- MCMClogit(Survived~., data = d.data) d.summ <- summary(d.res) d.summ #オッズ比 exp(d.summ$statistics[, "Mean"])
実行結果は下記の通りです。
実行結果
・・・
> d.res <- MCMClogit(Survived~., data = d.data)
>
> d.summ <- summary(d.res)
>
> d.summ
Iterations = 1001:11000
Thinning interval = 1
Number of chains = 1
Sample size per chain = 10000
1. Empirical mean and standard deviation for each variable,
plus standard error of the mean:
Mean SD Naive SE Time-series SE
(Intercept) 0.6775 0.2682 0.002682 0.012059
Class2nd -1.0367 0.1961 0.001961 0.008828
Class3rd -1.7935 0.1768 0.001768 0.008054
ClassCrew -0.8607 0.1596 0.001596 0.006971
SexFemale 2.4486 0.1384 0.001384 0.006279
AgeAdult -1.0540 0.2392 0.002392 0.010882
2. Quantiles for each variable:
2.5% 25% 50% 75% 97.5%
(Intercept) 0.1455 0.4992 0.6831 0.8630 1.2028
Class2nd -1.4185 -1.1683 -1.0331 -0.9027 -0.6626
Class3rd -2.1481 -1.9130 -1.7926 -1.6760 -1.4476
ClassCrew -1.1764 -0.9661 -0.8651 -0.7535 -0.5488
SexFemale 2.1543 2.3600 2.4525 2.5442 2.7110
AgeAdult -1.5275 -1.2144 -1.0573 -0.8904 -0.5907
>
> #オッズ比
> exp(d.summ$statistics[, "Mean"])
(Intercept) Class2nd Class3rd ClassCrew SexFemale AgeAdult
1.9690188 0.3546137 0.1663746 0.4228566 11.5715841 0.3485381
glm と同じような結果となりました。
最後に、それぞれの値の分布は下記のようになりました。


Windows上で Rust を使用
Rust はトレイト・パターンマッチ・アトリビュート等のモダンな言語機能を持ち、オブジェクト指向と純粋関数型のプログラミングスタイルをサポートしている、なかなか興味深いプログラミング言語です。
Rust 1.0 以降は http://fits.hatenablog.com/entry/2015/09/20/193605 を参照
環境構築
まずは Rust のビルド・実行環境を構築します。
といっても下記を実施するだけです。
(2) に関しては、Haskell Platform 2013.2.0.0 for Windows 等をインストールしてあれば、その中に含まれている mingw を代わりに使えば良いので必要ありません。
また、Rust 0.9 では GCC に依存しているので (2) が必要となっていますが、将来的には変わるかもしれません。
(1) Rust をインストール
Using Rust on Windows の installer リンクから rust-0.9-install.exe をダウンロード、インストールします。
(2) MinGW で gcc をインストール
http://sourceforge.net/projects/mingw/files/ から mingw-get-setup.exe をダウンロード、インストールします。
also install support for the graphical user interface のチェックを外してインストールした場合、「Quit」 ボタンを押下してインストールを終了する点にご注意ください。(この場合 「Continue」 ボタンは有効になりません)
コマンドラインでインストールする場合、mingw-get.exe (例 C:\MinGW\bin\mingw-get.exe) を使って gcc をインストールします。
MinGW へ gcc をインストール
> mingw-get install gcc
なお、Haskell Platform 2013.2.0.0 for Windows 等をインストールしてあれば、MinGW のインストールは不要です。
ビルドと実行
それでは下記のようなサンプルソースをビルドして実行してみます。
実行時の処理は main() 関数へ実装します。
sample.rs
fn main() {
let d1 = Data { name: ~"data", value: 10 };
let d2 = Data { name: ~"data", value: 10 };
let d3 = Data { name: ~"data", value: 0 };
let d4 = Data { name: ~"etc", value: 5 };
println!("d1 == d2 : {}", d1 == d2);
println!("d1 == d2 : {}", d1.eq(&d2));
println!("d1 == d3 : {}", d1 == d3);
println!("-----")
println!("{:?}", d1);
println!("{}", d1.to_str());
println!("-----")
println!("times = {}", d1.times(3));
println!("-----")
d1.printValue();
d3.printValue();
println!("-----")
let res = calc([d1, d2, d3, d4]);
println!("calc = {}", res);
}
fn calc(list: &[Data]) -> int {
list.iter().fold(1, |acc, v| acc * match v {
// name = "data" で value の値が 0 より大きい場合
&Data {name: ~"data", value: b} if b > 0 => b,
// それ以外
_ => 1
})
}
// Eq と ToStr トレイトを自動導出した struct 型の定義
#[deriving(Eq, ToStr)]
struct Data {
name: ~str,
value: int
}
// Data のメソッド定義と実装
impl Data {
fn printValue(&self) {
match self.value {
0 => println!("value: zero"),
a @ _ => println!("value: {}", a)
}
}
}
// トレイト定義
trait Sample {
fn get_value(&self) -> int;
fn times(&self, n: int) -> int {
self.get_value() * n
}
}
// Data へ Sample トレイトを実装
impl Sample for Data {
fn get_value(&self) -> int {
self.value
}
}
上記では deriving アトリビュートを使って Eq と ToStr トレイトを自動導出しています。
Eq の deriving で eq() メソッドが自動的に実装され == が使えるようになり、ToStr の deriving で to_str() メソッドが自動的に実装されます。
なお、struct 型(構造体)は {} で出力できないので、代わりに {:?} を使うか std::fmt::Default トレイトを実装する必要があります。
ちなみに、~ はポインタ(Owning pointers) で & はリファレンスです。
ビルド
まずは環境変数 PATH へ Rust と MinGW の bin ディレクトリのパスを設定しておきます。
環境変数 PATH の設定例1
set PATH=C:\Rust\bin;C:\MinGW\bin
MinGW の代わりに Haskell Platform 2013.2.0.0 for Windows を使う場合は下記のようになります。
環境変数 PATH の設定例2
set PATH=C:\Rust\bin;C:\Haskell Platform\2013.2.0.0\mingw\bin
rustc コマンドを使ってビルドを実施します。 ビルドに成功すると .exe ファイルが作成されます。
ビルド例
> rustc sample.rs
実行
ビルドで生成された .exe ファイルを実行します。
実行には Rust の bin ディレクトリ内の .dll ファイルを使いますので、Rust の bin は環境変数 PATH へ設定しておく必要があります。 (MinGW は基本的に不要です)
実行例
> sample.exe
d1 == d2 : true
d1 == d2 : true
d1 == d3 : false
-----
Data{name: ~"data", value: 10}
Data{name: data, value: 10}
-----
times = 30
-----
value: 10
value: zero
-----
calc = 100
今回使用したサンプルソースは http://github.com/fits/try_samples/tree/master/blog/20140223/
R で個体差のあるロジスティック回帰2 - MCMCglmm
前回 に続き、今回は個体差を考慮したロジスティック回帰を MCMCglmm で試してみます。
実は MCMCglmm の他にも MCMCpack の MCMChlogit や bayesm の rhierBinLogit 等といろいろ試そうとしてみたのですが、イマイチ使い方が分からなかったので今回は断念しました。
今回使用したサンプルソースは http://github.com/fits/try_samples/tree/master/blog/20140211/
はじめに
今回使用するパッケージを R へインストールしておきます。
install.packages("MCMCglmm")
(2) MCMCglmm を使った階層ベイズモデルによるロジスティック回帰(MCMCglmm 関数)
それでは、前回 と同じデータ (data7.csv) を使って推定と予測線のグラフ化を実施してみます。
MCMCglmm() による推定
MCMCglmm() 関数は glmmML() 関数とほぼ同じ使い方ができますが、
今回のケースでは下記の点が異なります。
- family に multinomial2 を指定する
- デフォルトで個体差 (units に該当) が考慮されるようなので特に id 列を指定する必要はない
デフォルトで詳細(処理状況)が出力されるので、下記では出力しないよう verbose = FALSE としています。
logiMcmcglmm.R
d <- read.csv('data7.csv') library(MCMCglmm) d.res <- MCMCglmm(cbind(y, N - y) ~ x, data = d, family = "multinomial2", verbose = FALSE) summary(d.res) ・・・
実行結果は下記のようになります。
実行結果
> d.res <- MCMCglmm(cbind(y, N - y) ~ x, data = d, family = "multinomial2", verbose = FALSE)
>
> summary(d.res)
Iterations = 3001:12991
Thinning interval = 10
Sample size = 1000
DIC: 667.8652
R-structure: ~units
post.mean l-95% CI u-95% CI eff.samp
units 6.988 3.898 10.58 517.4
Location effects: cbind(y, N - y) ~ x
post.mean l-95% CI u-95% CI eff.samp pMCMC
(Intercept) -4.2271 -6.3586 -2.5264 814.3 <0.001 ***
x 1.0144 0.6117 1.5237 800.3 <0.001 ***
---
Signif. codes: 0 '***' 0.001 '**' 0.01 '*' 0.05 '.' 0.1 ' ' 1
各パラメータの平均値 (post.mean) が で、前回の結果(glmmML)と近い値になりました。
ここで units は前回の個体差パラメータ の分散
に該当するようですので、標準偏差とするために平方根を取りました。
プログラム上で上記の結果を使う場合は、MCMCglmm() の結果から
(Intercept, x)の分布を
$Sol で、 (units)の分布を
$VCV で取得できます。
なお、実行する度に若干異なる値になるようなので、何らかのオプションを指定して調整する必要があるのかもしれません。
葉数と生存種子数
MCMCglmm() の結果を使って 前回 と同様にグラフ化してみます。
今回はパラメータの平均値(post.mean の値)を使わずに、事後最頻値 (posterior mode) というものを使って予測線を描画してみました。
事後最頻値は posterior.mode() 関数で取得できます。
logiMcmcglmm.R
・・・ # 生存確率の算出 calcProb <- function(x, b, r) 1.0 / (1.0 + exp(-1 * (b[1] + b[2] * x + r))) png("logiMcmcglmm_1.png") plot(d$x, d$y) xx <- seq(min(d$x), max(d$x), length = 100) # beta1 と beta2 の事後最頻値 beta <- posterior.mode(d.res$Sol) # s の事後最頻値 sd <- sqrt(posterior.mode(d.res$VCV)) lines(xx, max(d$N) * calcProb(xx, beta, 0), col="green") lines(xx, max(d$N) * calcProb(xx, beta, -1 * sd), col="blue") lines(xx, max(d$N) * calcProb(xx, beta, sd), col="blue") dev.off() ・・・
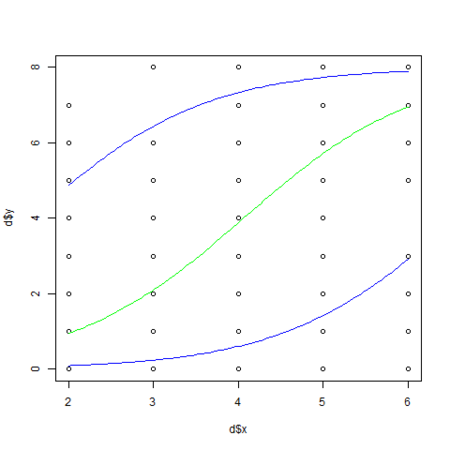
ついでに、前々回 のようにpredict() を使って予測線を描画しようとしてみましたが、今のところ predict.MCMCglmm() は新しいデータの指定に対応していないようです。
> predict(d.res, data.frame(x = xx)) Error in predict.MCMCglmm(d.res, data.frame(x = xx)) : sorry newdata not implemented yet
葉数  の種子数分布
の種子数分布
こちらも事後最頻値を使って、前回 と同様にグラフ化してみます。
logiMcmcglmm.R
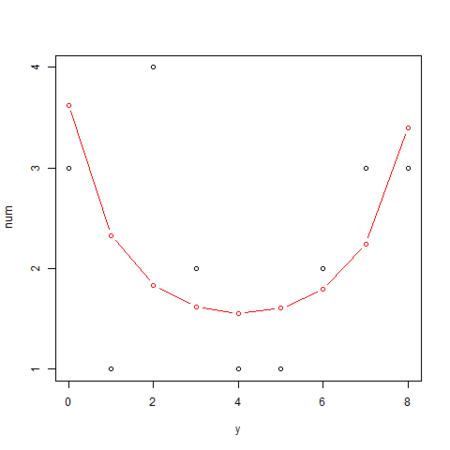
・・・ png("logiMcmcglmm_2.png") yy <- 0:max(d$N) plot(yy, table(d[d$x == 4,]$y), xlab="y", ylab="num") # 葉数 x を固定した場合の生存種子数 y の確率分布を算出 calcL <- function(ylist, xfix, n, b, s) sapply(ylist, function(y) integrate( f = function(r) dbinom(y, n, calcProb(xfix, b, r)) * dnorm(r, 0, s), lower = s * -10, upper = s * 10 )$value ) lines(yy, calcL(yy, 4, max(d$N), beta, sd) * length(d[d$x == 4,]$y), col="red", type="b") dev.off()
R で個体差のあるロジスティック回帰1 - glmmML
前回 のロジスティック回帰に続き、書籍 「 データ解析のための統計モデリング入門――一般化線形モデル・階層ベイズモデル・MCMC (確率と情報の科学) 」のサンプルを使って個体差を考慮したロジスティック回帰を GLMM と階層ベイズモデルで試してみます。
- (1) GLMM によるロジスティック回帰 (glmmML 関数)
- (2) MCMCglmm を使った階層ベイズモデルによるロジスティック回帰(MCMCglmm 関数)
ただし、今回は (1) だけで (2) に関しては次回に書く予定です。
サンプルソースは http://github.com/fits/try_samples/tree/master/blog/20140209/
はじめに
パッケージのインストール
今回使用するパッケージを R へインストールしておきます。
install.packages("glmmML")
データ
データは書籍のサポート web サイトから取得します。 今回は 7章「一般化線形混合モデル(GLMM)- 個体差のモデリング -」のサンプルデータを使います。
データ data7.csv
"N","y","x","id" 8,0,2,1 8,1,2,2 8,2,2,3 ・・・
データ内容は下記の通りです。
| 項目 | 内容 |
|---|---|
| N | 調査種子数 |
| y | 生存種子数 |
| x | 植物の葉数 |
| id | 個体のID |
葉数 の値が 2 ~ 6 までとなっており、
葉数毎に 20 個体ずつの生存種子数
をサンプリングしたデータとなっています。 (合計 100 個体)
二項分布の性質と過分散
ところで、二項分布の性質として下記があります。
今回のケースでは n が調査種子数(= 8)、p が生存確率です。
- (1) 平均(期待値とするのが適切かも)が
- (2) 分散が
ここで、今回のデータは下記のようになっています。
葉数 4(x = 4)場合の平均と分散
> mean(d[d$x == 4,]$y) [1] 4.05 > var(d[d$x == 4,]$y) [1] 8.365789
np = 4.05 とすると p = 4.05 / 8 = 約 0.5 となり、二項分布として期待される分散は np(1 - p) = 8 × 0.5 × (1 - 0.5) = 2 で、実際の分散 8.36 の方が明らかに大きい値となっています。 (過分散)
二項分布として考えると、原因不明な個体差等による効果を考慮した統計モデルを使ったロジスティック回帰が必要となり、GLMM や階層ベイズを使用するという流れとなるようです。
(1) GLMM によるロジスティック回帰 (glmmML 関数)
それでは glmmML() 関数を使った推定値の算出とグラフ化を実施してみます。
推定方法
個体差を考慮するため、GLMM では個体差のパラメータ を追加した線形予測子
を使う事になるようです。
ここで、 は生存確率です。
そして、 が正規分布の確率分布に従っていると仮定し、
確率密度関数 を加味して
を積分した
尤度 に対して
全データの積 の対数尤度
が最大になるようなパラメータ
の最尤推定値を求めます。
なお、 は二項分布の確率密度関数で、
は個体差
のばらつき(標準偏差)です。
glmmML() による推定
glmmML() 関数へ指定するオプションは、前回 の glm() とほとんど同じですが 、下記の点が異なります。
- 個体差のパラメータ
部分を cluster オプションで指定
今回は個体毎に異なる値が設定されている id 列を cluster オプションへ指定します。
logiGlmmML.R
d <- read.csv('data7.csv') library(glmmML) d.res <- glmmML(cbind(y, N - y) ~ x, data = d, family = binomial, cluster = id) summary(d.res) ・・・
glmmML() による推定結果は下記の通りです。
実行結果
Call: glmmML(formula = cbind(y, N - y) ~ x, family = binomial, data = d, cluster = id)
coef se(coef) z Pr(>|z|)
(Intercept) -4.190 0.8777 -4.774 1.81e-06
x 1.005 0.2075 4.843 1.28e-06
Scale parameter in mixing distribution: 2.408 gaussian
Std. Error: 0.2202
LR p-value for H_0: sigma = 0: 2.136e-55
Residual deviance: 269.4 on 97 degrees of freedom AIC: 275.4
と推定されました。
なお、glmmML() の結果から、
の値は
$coefficients で、 の値は
$sigma で取得できます。
葉数と生存種子数
推定したパラメータを使って葉数 x と生存種子数 y の予測線をグラフ化する処理は下記のようになります。
生存種子数の予測値は 調査種子数(= max(d$N) = 8)× 生存確率 で算出し、
生存確率 は
の式を元に算出します。
logiGlmmML.R
・・・ # 生存確率の算出 calcProb <- function(x, b, r) 1.0 / (1.0 + exp(-1 * (b[1] + b[2] * x + r))) png("logiGlmmML_1.png") # x と y をプロット plot(d$x, d$y) # x の範囲(2 ~ 6)を 100分割 xx <- seq(min(d$x), max(d$x), length = 100) beta <- d.res$coefficients # 個体差 r = 0 の葉数と生存種子数との予測線 lines(xx, max(d$N) * calcProb(xx, beta, 0), col="green") # 個体差 r = -2.408 の葉数と生存種子数との予測線 lines(xx, max(d$N) * calcProb(xx, beta, -1 * d.res$sigma), col="blue") # 個体差 r = 2.408 の葉数と生存種子数との予測線 lines(xx, max(d$N) * calcProb(xx, beta, d.res$sigma), col="blue") dev.off() ・・・
個体差 r = 0 (個体差なし)とした場合の予測線を緑色で、 r = ±2.408 の予測線を青色で描画しています。

葉数  の生存種子数分布
の生存種子数分布
次に、葉数 x が 4 の場合の生存種子数 y と個体数の分布と予測線をグラフ化する処理は下記のようになります。
glmmML() の結果から、x = 4 の場合の y の確率分布を算出し、x = 4 のサンプル数(= 20)を乗算して予測線を描画しています。
y の確率分布は、0 ~ 8 のそれぞれの値に対して で算出します。 (二項分布 × 正規分布 を個体差
で積分)
下記では sapply() と integrate() 関数を使って上記の算出処理を実装しています。
なお、lower と upper オプションを使って積分範囲を から
としています。
logiGlmmML.R
・・・ png("logiGlmmML_2.png") # 0 ~ 8 yy <- 0:max(d$N) # 生存種子数 y と個体数の分布 plot(yy, table(d[d$x == 4,]$y), xlab="y", ylab="num") # 葉数 x を固定した場合の生存種子数 y の確率分布を算出 calcL <- function(ylist, xfix, n, b, s) sapply(ylist, function(y) integrate( f = function(r) dbinom(y, n, calcProb(xfix, b, r)) * dnorm(r, 0, s), lower = s * -10, upper = s * 10 )$value ) # 予測線 lines(yy, calcL(yy, 4, max(d$N), beta, d.res$sigma) * length(d[d$x == 4,]$y), col="red", type="b") dev.off()

今回はここまで。 MCMCglmm を使った階層ベイズは次回。
Gradle で Jetty9 を使用
今のところ Gradle 標準の jetty プラグインでは Jetty6 しか使えないようなので、Servlet 3.0 を使いたいケースでは不便です。
この場合、build.gradle で Jetty9 の起動処理を自前で実装する手も考えられますが、Gradle Jetty Plugin for Eclipse Jetty (gradle-jetty-eclipse-plugin) を使った方が簡単だと思います。 (他にも gradle-jetty9-plugin というのもあります )
gradle-jetty-eclipse-plugin を使うと gradle jettyEclipseRun で Jetty 9.0.6 を起動できます。
gradle-jetty-eclipse-plugin の使い方
使用するには、build.gradle へ下記の設定を追加するだけです。
- gradle-jetty-eclipse-plugin を使用するための buildscript を定義
jettyEclipseを apply plugin する
gradle-jetty-eclipse-plugin を使用するための build.gradle 設定
apply plugin: 'jettyEclipse' buildscript { repositories { jcenter() maven { url 'http://dl.bintray.com/khoulaiz/gradle-plugins' } } dependencies { classpath (group: 'com.sahlbach.gradle', name: 'gradle-jetty-eclipse-plugin', version: '1.9.+') } }
なお、jcenter() の部分は Jetty9 のモジュールを取得できるリポジトリであればよいようなので mavenCentral() でも問題ないようです。
Jetty9 の実行
それでは @WebServlet アノテーションを使った単純な Servlet を実行してみます。
サンプルソースは http://github.com/fits/try_samples/tree/master/blog/20140203/ に配置しています。
まずは build.gradle です。
gradle-jetty-eclipse-plugin の利用設定を追加し、providedCompile で Servlet 3.0 の API を指定しました。
build.gradle
apply plugin: 'jettyEclipse' buildscript { repositories { jcenter() maven { url 'http://dl.bintray.com/khoulaiz/gradle-plugins' } } dependencies { classpath (group: 'com.sahlbach.gradle', name: 'gradle-jetty-eclipse-plugin', version: '1.9.+') } } repositories { mavenCentral() } dependencies { providedCompile 'javax.servlet:javax.servlet-api:3.0.1' }
Servlet は "sample" という文字を出力するだけの単純な処理を実装しました。
SampleServlet.java
package fits.sample; import java.io.IOException; import javax.servlet.ServletException; import javax.servlet.annotation.WebServlet; import javax.servlet.http.HttpServlet; import javax.servlet.http.HttpServletRequest; import javax.servlet.http.HttpServletResponse; @WebServlet(urlPatterns = {"/sample"}) public class SampleServlet extends HttpServlet { @Override protected void doGet(HttpServletRequest req, HttpServletResponse res) throws ServletException, IOException { res.getWriter().print("sample"); } }
それでは gradle jettyEclipseRun で実行してみます。
ちなみに、標準 jetty プラグインのような jettyRun と jettyRunWar のような区別はありません。
実行
> gradle jettyEclipseRun :compileJava :processResources UP-TO-DATE :classes :war :jettyEclipseRun Empty contextPath !RequestLog Hit <ENTER> to reload the webapp. Hit r + <ENTER> to rebuild and reload the webapp. Hit R + <ENTER> to rebuild the webapp without reload > Building 80% > :jettyEclipseRun > Running at http://localhost:8080/
これで Jetty9 が起動し、http://localhost:8080/sample へ接続すると "sample" という文字が返ってくるはずです。
Commons OGNL でマッピング・フィルタリング・畳み込み
以前、Java でマッピング・フィルタリング・畳み込みを試しましたが、 今回は Commons OGNL を使って OGNL 式によるマッピング・フィルタリング・畳み込みを Groovy で試してみました。
サンプルソースは http://github.com/fits/try_samples/tree/master/blog/20140118/
はじめに
Commons OGNL ではマッピングとフィルタリング用の式は用意されてますが、今のところ畳み込みは用意されていないようです。 (一応、畳み込みはラムダ式 :[ e ] で実装できました)
| 処理 | 機能名 | OGNL式 |
|---|---|---|
| マッピング | Projection | e1.{ e2 } |
| フィルタリング | Selection | e1.{? e2 } |
| 畳み込み | - | - |
Selection には、マッチした最初の要素を返す e1.{^ e2 } や最後の要素を返す e1.{$ e2 } のようなバリエーションも用意されています。
サンプルで使用するモデルクラス
今回のサンプルでは下記のようなモデルクラスを使いました。
Order.groovy
import groovy.transform.* @CompileStatic class Order { List<OrderLine> lines = [] } @CompileStatic @Immutable class OrderLine { String code BigDecimal price = 0 }
groovyc でコンパイルしておきます。
コンパイル
> groovyc Order.groovy
マッピング・フィルタリング
OGNL 式を使ったマッピング・フィルタリング処理を Groovy スクリプトで試してみます。
まず、@Grab を使って普通に Ognl.getValue(<OGNL式>, <オブジェクト>) を実行しようとすると下記のようなエラーが発生します。
エラー内容
java.lang.IllegalArgumentException: Javassist library is missing in classpath! Please add missed dependency!
at org.apache.commons.ognl.OgnlRuntime.getCompiler(OgnlRuntime.java:210) ・・・
Caused by: java.lang.ClassNotFoundException: Unable to resolve class: javassist.ClassPool
at org.apache.commons.ognl.OgnlRuntime.classForName(OgnlRuntime.java:665)
・・・
これは、デフォルトで使用される DefaultClassResolver が SystemClassLoader から javassist.ClassPool をロードしようとする事に起因するようで、Servlet 等の Java EE Web アプリケーションで実行する際にも同様のエラーが発生します。 (war ファイル内の JAR からロードするような場合)
エラー内容から CLASSPATH に javassist.jar が含まれていないように思ってしまいますが、そうではありません。
DefaultClassResolver に原因があるので、自前の ClassResolver を使って作成した Context を使うようにすればエラーを回避できます。
map_filter_sample.groovy
@GrabResolver('http://repository.apache.org/snapshots/') @Grab('org.apache.commons:commons-ognl:4.0-SNAPSHOT') import org.apache.commons.ognl.Ognl import org.apache.commons.ognl.ClassResolver def data = new Order() data.lines << new OrderLine('1', 100) data.lines << new OrderLine('2', 200) data.lines << new OrderLine('3', 300) try { // (1) DefaultClassResolver が原因でエラーが発生 println Ognl.getValue('lines.{? #this.code == "2" }', data) } catch (e) { println '(1) ' + e //e.printStackTrace() } println '-----------------------' // エラー回避のために自前の ClassResolver を使って Context 作成 def ctx = Ognl.createDefaultContext(null, { String className, Map<String, Object> context -> Class.forName(className) } as ClassResolver) // (2) マッピング println '(2) ' + Ognl.getValue('lines.{ #this.price > 100 }', ctx, data) // (3) フィルタリング println '(3) ' + Ognl.getValue('lines.{? #this.price > 100 }', ctx, data) // (4) マッチした最初の要素 println '(4) ' + Ognl.getValue('lines.{^ #this.price > 100 }', ctx, data) // (5) マッチした最後の要素 println '(5) ' + Ognl.getValue('lines.{$ #this.price > 100 }', ctx, data) // (6) OGNL式をコンパイルして使用 def exprNode = Ognl.compileExpression(ctx, null, 'lines.{? #this.code not in {"2", "4"} }') println '(6) ' + Ognl.getValue(exprNode, data)
実行結果は下記の通りです。
実行結果
> groovy map_filter_sample.groovy (1) java.lang.IllegalArgumentException: Javassist library is missing in classpath! Please add missed dependency! ----------------------- (2) [false, true, true] (3) [OrderLine(2, 200), OrderLine(3, 300)] (4) [OrderLine(2, 200)] (5) [OrderLine(3, 300)] (6) [OrderLine(1, 100), OrderLine(3, 300)]
畳み込み
OGNL のラムダ記法 :[ 処理 ] を使って畳み込みを試しに実装してみました。
OGNL のラムダは引数を一つしか取れないようなので(ラムダ内の #this にバインドされます)、リストを使ってラムダ #fold へ引数 {<処理>, <値>, <要素リスト>} を渡すようにしています。
更に、OrderLine の price を合計するための処理もラムダで定義して (:[ #this[0] + #this[1].price ])、#fold へ渡しています。
fold_sample.groovy
・・・ // 畳み込みの OGNL 式 def foldOgnl = ''' #fold = :[ #this[2].size() > 0 ? #fold({ #this[0], #this[0]({ #this[1], #this[2][0] }), #this[2].subList(1, #this[2].size()) }) : #this[1] ], #fold({ :[ #this[0] + #this[1].price ], 0, lines }) ''' // 0 + 100 + 200 + 300 println Ognl.getValue(foldOgnl, ctx, data)
実行結果
> groovy fold_sample.groovy 600
なお、合計を計算するだけなら以下のようにした方がシンプルです。
println Ognl.getValue('#v = 0, lines.{ #v = #v + #this.price }, #v', ctx, data)
並列実行時の注意点
最後に、並列処理で実行する際の注意点です。
問題確認のために、下記 3種類の処理を 50回並列でそれぞれ行ってみて OGNL の処理結果が正しかったかどうかを true・false で返すようにしてみます。
- (1) 文字列の OGNL 式で Context を再利用して getValue を実行
- (2) 文字列の OGNL 式で Context を都度作成して getValue を実行
- (3) compileExpression した結果で getValue を実行
parallel_sample.groovy
@GrabResolver('http://repository.apache.org/snapshots/') @Grab('org.apache.commons:commons-ognl:4.0-SNAPSHOT') import org.apache.commons.ognl.Ognl import org.apache.commons.ognl.ClassResolver import groovyx.gpars.* def createData = { i -> def data = new Order() data.lines << new OrderLine('1', i) data.lines << new OrderLine('2', i + 1) data.lines << new OrderLine('3', i + 2) data } def sum = { data -> data.lines.inject(0){ acc, val -> acc + val.price } } def printResult = { it.groupBy().each { k, v -> println "${k} ${v.size()}" } } def createContext = { Ognl.createDefaultContext(null, { String className, Map<String, Object> context -> Class.forName(className) } as ClassResolver) } def ctx = createContext() // OGNL式をコンパイル def exprNode = Ognl.compileExpression(ctx, null, '#v = 0, lines.{ #v = #v + #this.price }, #v') def count = 50 GParsPool.withPool(20) { // (1) 文字列の OGNL 式で並列処理(Context再利用) def res1 = (0..<count).collectParallel { def d = createData(it) try { // 稀に NoSuchPropertyException: Order.price が発生するため try-catch sum(d) == Ognl.getValue('#v = 0, lines.{ #v = #v + #this.price }, #v', ctx, d) } catch (e) { println e false } } // (2) 文字列の OGNL 式で並列処理(Context毎回作成) def res2 = (0..<count).collectParallel { def d = createData(it) sum(d) == Ognl.getValue('#v = 0, lines.{ #v = #v + #this.price }, #v', createContext(), d) } // (3) compileExpression 結果で並列処理 def res3 = (0..<count).collectParallel { def d = createData(it) sum(d) == Ognl.getValue(exprNode, d) } println '----- (1) 文字列の OGNL 式で並列処理(Context再利用)------' printResult res1 println '----- (2) 文字列の OGNL 式で並列処理(Context毎回作成) ---' printResult res2 println '----- (3) compileExpression 結果で並列処理 ----------------' printResult res3 }
処理結果は以下のようになり、(1) のケースで問題が発生しています。
実行結果
> groovy parallel_sample.groovy org.apache.commons.ognl.NoSuchPropertyException: Order.price ----- (1) 文字列の OGNL 式で並列処理(Context再利用)------ true 36 false 14 ----- (2) 文字列の OGNL 式で並列処理(Context毎回作成) --- true 50 ----- (3) compileExpression 結果で並列処理 ---------------- true 50
これは (1) のように Context を再利用すると #v や #this を並列処理間で共有してしまう事が原因と考えられます。
そのため、並列処理で実行する際は Context をその都度作成するか、compileExpression した結果を使う事になると思います。 (他の方法もあるかもしれません)