Apache Spark でロジスティック回帰
以前 ※ に R や Julia で試したロジスティック回帰を Apache Spark の MLlib (Machine Learning Library) を使って実施してみました。
サンプルソースは http://github.com/fits/try_samples/tree/master/blog/20150427/
※「 R でロジスティック回帰 - glm, MCMCpack 」、「 Julia でロジスティック回帰-glm 」
はじめに
R の時と同じデータを使いますが、ヘッダー行を削除しています。(「R でロジスティック回帰 - glm, MCMCpack」 参照)
データ data4a.csv
8,1,9.76,C 8,6,10.48,C 8,5,10.83,C ・・・
データ内容は以下の通り。個体 i それぞれにおいて 「 個の観察種子のうち生きていて発芽能力があるものは
個」 となっています。
| 項目 | 内容 |
|---|---|
| N | 観察種子数 |
| y | 生存種子数 |
| x | 植物の体サイズ |
| f | 施肥処理 (C: 肥料なし, T: 肥料あり) |
体サイズ x と肥料による施肥処理 f が種子の生存する確率(ある個体 i から得られた種子が生存している確率)にどのように影響しているかをロジスティック回帰で解析します。
MLlib によるロジスティック回帰
今回は org.apache.spark.mllib.classification.LogisticRegressionWithLBFGS を使用します。
LogisticRegressionWithLBFGS について
LogisticRegressionWithLBFGS で以前と同様のロジスティック回帰を実施するには以下が必要です。
setInterceptで true を設定
この値が false (デフォルト値) の場合、結果の intercept 値が 0 になります。
なお、今回のように二項分布を使う場合は numClasses の値を変更する必要はありませんが (デフォルト値が 2 のため)、応答変数が 3状態以上の多項分布を使う場合は setNumClasses で状態数に応じた値を設定します。
LabeledPoint について
LogisticRegressionWithLBFGS へ与えるデータは LabeledPoint で用意します。
R や Julia では 応答変数 ~ 説明変数1 + 説明変数2 + ・・・ のように応答変数と説明変数を指定しましたが、LabeledPoint では下記のようにメンバー変数で表現します。
| メンバー変数 | 応答変数・説明変数 |
|---|---|
| label | 応答変数 |
| features | 説明変数 |
値は Double とする必要がありますので、f 項目のような文字列値は数値化します。
更に、二項分布を使う場合 (numClasses = 2) は応答変数の値が 0 か 1 でなければなりません。
LabeledPoint への変換例
例えば、以下のようなデータを応答変数 y 項目、説明変数 x と f 項目の LabeledPoint へ変換する場合
変換前のデータ (N = 8, y = 6)
8,6,10.48,C
次のようになります。
変換後のデータイメージ
LabeledPoint(label: 1.0, features: Vector(10.48, 0.0)) LabeledPoint(label: 1.0, features: Vector(10.48, 0.0)) LabeledPoint(label: 1.0, features: Vector(10.48, 0.0)) LabeledPoint(label: 1.0, features: Vector(10.48, 0.0)) LabeledPoint(label: 1.0, features: Vector(10.48, 0.0)) LabeledPoint(label: 1.0, features: Vector(10.48, 0.0)) LabeledPoint(label: 0.0, features: Vector(10.48, 0.0)) LabeledPoint(label: 0.0, features: Vector(10.48, 0.0))
8個(N)の中で 6個(y)生存していたデータのため、
label (応答変数) の値が 1.0 (生存) のデータ 6個と 0.0 のデータ 2個へ変換します。
ちなみに、f 項目の値が C の場合は 0.0、T の場合は 1.0 としています。
実装
実装してみると以下のようになります。
LogisticRegression.scala
import org.apache.spark.SparkContext import org.apache.spark.mllib.classification.LogisticRegressionWithLBFGS import org.apache.spark.mllib.regression.LabeledPoint import org.apache.spark.mllib.linalg.Vectors object LogisticRegression extends App { // f項目の値を数値へ変換 val factor = (s: String) => s match { case "C" => 0 case _ => 1 } val sc = new SparkContext("local", "LogisticRegression") // データの準備 (100行のデータ -> 800個の LabeledPoint) val rdd = sc.textFile(args(0)).map(_.split(",")).flatMap { d => val n = d(0).toInt val x = d(1).toInt // 説明変数の値 val v = Vectors.dense(d(2).toDouble, factor(d(3))) // 応答変数が 1 のデータ x 個と 0 のデータ n - x 個を作成 List.fill(x)( LabeledPoint(1, v) ) ++ List.fill(n -x)( LabeledPoint(0, v) ) } // ロジスティック回帰の実行 val res = new LogisticRegressionWithLBFGS() // .setNumClasses(2) //省略可 .setIntercept(true) .run(rdd) println(res) }
ビルド
以下のような Gradle ビルド定義ファイルを使って実行します。
build.gradle
apply plugin: 'scala' apply plugin: 'application' mainClassName = 'LogisticRegression' repositories { jcenter() } dependencies { compile 'org.scala-lang:scala-library:2.11.6' compile('org.apache.spark:spark-mllib_2.11:1.3.1') { // ログ出力の抑制 exclude module: 'slf4j-log4j12' } // ログ出力の抑制 runtime 'org.slf4j:slf4j-nop:1.7.12' } run { if (project.hasProperty('args')) { args project.args.split(' ') } }
不要な WARN ログ出力を抑制するため以下のファイルも用意しました。
src/main/resources/log4j.properties
log4j.rootLogger=off
実行
実行結果は以下の通りです。
実行結果
> gradle run -Pargs=data4a.csv :clean :compileJava UP-TO-DATE :compileScala :processResources :classes :run (weights=[1.952347703282676,2.021401680901667], intercept=-19.535421113192506) BUILD SUCCESSFUL
以前に実施した R の結果 (Estimate の値) とほとんど同じ値になっています。
R の glm 関数による結果
Coefficients:
Estimate Std. Error z value Pr(>|z|)
(Intercept) -19.5361 1.4138 -13.82 <2e-16 ***
x 1.9524 0.1389 14.06 <2e-16 ***
fT 2.0215 0.2313 8.74 <2e-16 ***
Julia でロジスティック回帰 - glm
前回 に続き、今回も Julia で GLM を実施します。
今回は 「R でロジスティック回帰 - glm, MCMCpack」 のロジスティック回帰(GLM)を Julia で実装してみました。
サンプルソースは http://github.com/fits/try_samples/tree/master/blog/20150309/
はじめに
GLM 等のパッケージは 前回 と同じものを使用します。
データは R で試した時のものをそのまま使います。(「R でロジスティック回帰 - glm, MCMCpack」 参照)
データ data4a.csv
N,y,x,f 8,1,9.76,C 8,6,10.48,C 8,5,10.83,C ・・・
データの内容は以下の通りで、個体 i それぞれにおいて 「 個の観察種子のうち生きていて発芽能力があるものは
個」 となっています。
| 項目 | 内容 |
|---|---|
| N | 観察種子数 |
| y | 生存種子数 |
| x | 植物の体サイズ |
| f | 施肥処理 (C: 肥料なし, T: 肥料あり) |
体サイズ x と肥料による施肥処理 f が種子の生存する確率(ある個体 i から得られた種子が生存している確率)にどのように影響しているかをロジスティック回帰で解析します。
GLM によるロジスティック回帰
Julia の GLM パッケージを使う場合の注意点は下記の通りです。
- (1) Binomial (二項分布) を使う場合は応答変数(下記の y に該当)の値が 0.0 ~ 1.0 内でなければならない
- (2) readtable ではカテゴリ型変数 (R の因子型に該当) 化を実施しない
(1) への対応として、y を N で割った値 yn を応答変数(目的変数)として使いました。
今のところ d[:yn] = d[:y] / d[:N] とは書けないようなので map 関数を使っています。
R の read.csv 関数等では f のような項目は因子型になると思いますが、Julia の readtable では (2) のようにカテゴリ型変数とはならず単なる文字列の配列となります。 (d[:f] の型は DataArrays.DataArray{UTF8String,1} になる)
glm 関数で文字列の変数は使えないので、カテゴリ型変数への変換が必要となります。
DataFrames パッケージにはカテゴリ型への変換関数 pool が用意されているので、下記では f 項目の値を pool 関数で変換し ff へ設定しています。 (d[:ff] の型は DataArrays.PooledDataArray{UTF8String,UInt8,1} になる)
logisticGlm.jl
using DataFrames, GLM d = readtable("data4a.csv") # (1) 生存率 y / N の算出 d[:yn] = map(x -> d[:y][x] / d[:N][x], 1:nrow(d)) # (2) カテゴリ型へ変換 (DataArrays.PooledDataArray{UTF8String,UInt8,1} になる) d[:ff] = pool(d[:f]) # 以下でも可 (ただし、DataArrays.PooledDataArray{UTF8String,UInt32,1} になる) # d[:ff] = convert(PooledDataArray, d[:f]) res = glm(yn~x + ff, d, Binomial()) println(res)
実行結果は以下のようになりました。 (R で実施したときと同じ Estimate 値)
実行結果
> julia logisticGlm.jl
DataFrames.DataFrameRegressionModel{GLM.GeneralizedLinearModel{GLM.GlmResp{Array{Float64,1},Distributions.Binomial,GLM.LogitLink},GLM.DensePredChol{Float64}},Float64}:
Coefficients:
Estimate Std.Error z value Pr(>|z|)
(Intercept) -19.5361 3.99861 -4.88572 <1e-5
x 1.95241 0.392777 4.97077 <1e-6
ff - T 2.02151 0.654152 3.09027 0.0020
ちなみに、d[:f] の内容と pool・convert 関数の適用結果は以下のようになります。
julia> d[:f]
100-element DataArrays.DataArray{UTF8String,1}:
"C"
"C"
・・・
"T"
"T"
julia> pool(d[:f])
100-element DataArrays.PooledDataArray{UTF8String,UInt8,1}:
"C"
"C"
・・・
"T"
"T"
julia> convert(PooledDataArray, d[:f])
100-element DataArrays.PooledDataArray{UTF8String,UInt32,1}:
"C"
"C"
・・・
"T"
"T"
PooledDataArray
PooledDataArray の水準名の一覧は levels 関数か pool フィールドを使って取得できます。
julia> levels(d[:ff])
2-element Array{UTF8String,1}:
"C"
"T"
julia> d[:ff].pool
2-element Array{UTF8String,1}:
"C"
"T"
水準名と数値のマッピングは levelsmap 関数で取得できます。
julia> levelsmap(d[:ff])
Dict{UTF8String,Int64} with 2 entries:
"T" => 2
"C" => 1
また、refs フィールドで各データに割り当てられた数値を取得できます。
julia> d[:ff].refs
100-element Array{UInt8,1}:
0x01
0x01
・・・
0x02
0x02
予測線の描画
次に glm の結果 (DataFrameRegressionModel) を使って予測腺を描画します。
glm の結果では、「肥料あり "T" => 1、肥料なし "C" => 0」 の扱いになっているので、predict する際に肥料ありは 1 を肥料なしは 0 の配列を渡します。
predict の結果は生存率 yn の予測値なので、グラフへ描画する際に観察種子数 N の最大値 (今回は全て 8 なので最大値とする必要はない) を乗算しています。
なお、rep 関数を使うと指定の配列を指定回数繰り返した配列を作成できます。
logisticGlm_draw.jl
using DataFrames, GLM, Gadfly ・・・ res = glm(yn~x + ff, d, Binomial()) # x の最小値 7.66 ~ 最大値 12.44 まで 0.1 刻みのデータを用意 xx = [minimum(d[:x]):0.1:maximum(d[:x])] # 肥料あり "T" の予測値を算出 rt = predict(res, DataFrame(n = rep([1], length(xx)), x = xx, ff = rep([1], length(xx)))) # 肥料なし "C" の予測値を算出 rc = predict(res, DataFrame(n = rep([1], length(xx)), x = xx, ff = rep([0], length(xx)))) p = plot( layer(d, x = "x", y = "y", color = "f", Geom.point), # 肥料あり "T" の予測線を赤で描画 layer(x = xx, y = maximum(d[:N]) * rt, Geom.line, Theme(default_color = color("red"))), # 肥料なし "C" の予測線を緑で描画 layer(x = xx, y = maximum(d[:N]) * rc, Geom.line, Theme(default_color = color("green"))) ) draw(PNG("logisticGlm_draw.png", 500px, 400px), p)
実行結果
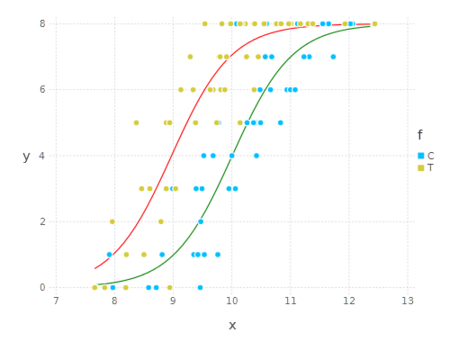
なお、rt と rc の算出処理を PooledDataArray を使って実装すると以下のようになります。
PooledDataArray では "T" => 2, "C" => 1 となっているため、"T" => 1, "C" => 0 でそれぞれ predict するように <PooledDataArray変数>.refs - 1 としています。
・・・ xx = [minimum(d[:x]):0.1:maximum(d[:x])] # 肥料あり "T" のカテゴリ型データを作成 (d[:ff] の水準を使用) ft = PooledDataArray(rep([utf8("T")], length(xx)), d[:ff].pool) # 肥料なし "C" のカテゴリ型データを作成 (d[:ff] の水準を使用) fc = PooledDataArray(rep([utf8("C")], length(xx)), d[:ff].pool) # 肥料あり "T" の予測値を算出 rt = predict(res, DataFrame(n = rep([1], length(xx)), x = xx, ff = ft.refs - 1)) # 肥料なし "C" の予測値を算出 rc = predict(res, DataFrame(n = rep([1], length(xx)), x = xx, ff = fc.refs - 1)) ・・・
Julia でポアソン回帰 - glm
以前、「R でポアソン回帰 - glm, MCMCpack」 にて試した GLM によるポアソン回帰を Julia で実施してみました。
なお、Julia は開発中の v0.4.0 を使用しました。
サンプルソースは http://github.com/fits/try_samples/tree/master/blog/20150223/
はじめに
今回は下記のパッケージを使用しますので、予めインストールしておきます。
| パッケージ名 | 用途 |
|---|---|
| DataFrames | csv ファイルの読み込み。データフレーム化 |
| GLM | GLM の実施 |
| Gadfly | グラフの描画 |
| Cairo | PNGファイルへのグラフ描画 |
パッケージのインストール例
> julia ・・・ julia> Pkg.add("DataFrames") ・・・ julia> Pkg.add("GLM") ・・・ julia> Pkg.add("Gadfly") ・・・ julia> Pkg.add("Cairo") ・・・
ポアソン回帰を試すデータは 以前 に使ったものをそのまま使用します。
データ data3a.csv
y,x,f 6,8.31,C 6,9.44,C 6,9.5,C 12,9.07,C 10,10.16,C ・・・
データ内容は下記の通りです。
今回は、体サイズ x が種子数 y にどのように影響しているかをポアソン回帰で解析します。
| 項目 | 内容 |
|---|---|
| y | 種子数 |
| x | 植物の体サイズ |
| f | 施肥処理 |
GLM によるポアソン回帰
y ~ x のモデルを使ってポアソン回帰を実施します。
以前、R で実施したものと同じポアソン回帰を行うには、glm 関数へ Poisson() と LogLink() (リンク関数)を指定します。
poissonGlm.jl
using DataFrames, GLM d = readtable("data3a.csv") res = glm(y ~ x, d, Poisson(), LogLink()) println(res)
実行結果は、以下のように R の結果とほぼ同じになりました。 (前回 の結果を参照)
今回は Julia の開発バージョンを使ったため deprecated syntax の WARNING がいくつか出力されました。
実行結果
> julia poissonGlm.jl
・・・
Coefficients:
Estimate Std.Error z value Pr(>|z|)
(Intercept) 1.29172 0.363686 3.55174 0.0004
x 0.0756619 0.0356042 2.12509 0.0336
この結果より、平均種子数の予測値を求める関数は となります。
予測線の描画1
次に、ポアソン回帰の結果を使って種子数 y の予測線を描画し PNG ファイルへ出力してみます。
まず、種子数 y の予測値を算出するために、data3a.csv における x の最小値 7.19 から最大値 12.4 までを 0.1 刻みにした配列 xx を作成しました。
coef 関数を使えば glm の結果から Coefficients の Estimate 値を配列として取得できるので、これと xx を使って種子数 y の予測値を算出できます。
plot と layer を組み合わせる事で複数のグラフを重ねて描画できます。
poissonGlm_draw1.jl
using DataFrames, GLM, Gadfly d = readtable("data3a.csv") res = glm(y ~ x, d, Poisson(), LogLink()) # x の最小値 7.19 ~ 最大値 12.4 まで 0.1 刻みのデータを用意 xx = [minimum(d[:x]):0.1:maximum(d[:x])] # 種子数 y の予測値を算出 yy = exp(coef(res)[1] + coef(res)[2] * xx) # グラフの描画 p = plot( # data3a.csv の内容を描画 layer(d, x = "x", y = "y", color = "f", Geom.point), # 予測線の描画 layer(DataFrame(x = xx, y = yy), x = "x", y = "y", Geom.line) ) # PNG 形式で出力 draw(PNG("poissonGlm_draw1.png", 500px, 400px), p)

ここで、xx は以下のような内容です。
xx の内容
53-element Array{Float64,1}:
7.19
7.29
・・・
12.29
12.39
予測線の描画2
最後に、種子数 y の予測値を predict 関数で算出し、同様のグラフを描画します。
predict を使うには x の値だけでは無く (Intercept) の Estimate 値へ乗算する値も必要になるので、下記では DataFrame (下記の nd) へ項目 n (常に 1) として用意しました。
predict 内では のような計算が実施されます。
poissonGlm_draw2.jl
・・・ res = glm(y ~ x, d, Poisson(), LogLink()) # 7.19 ~ 12.4 まで 0.1 刻みのデータを用意 xx = [minimum(d[:x]):0.1:maximum(d[:x])] # predict へ渡すデータを作成 nd = DataFrame(n = [1 for i = 1:length(xx)], x = xx) # predict で種子数 y の予測値を算出 yy = predict(res, nd) p = plot( # data3a.csv の内容を描画 layer(d, x = "x", y = "y", color = "f", Geom.point), # 予測線の描画 layer(DataFrame(x = xx, y = yy), x = "x", y = "y", Geom.line) ) draw(PNG("poissonGlm_draw2.png", 500px, 400px), p)

R でロジスティック回帰とオッズ比の算出 - glm, MCMClogit
以前、glm・MCMCmetrop1R 関数でロジスティック回帰を試みましたが、今回はその時に利用を断念した MCMCpack の MCMClogit 関数を使ってロジスティック回帰を行います。
題材は、書籍 「 データサイエンティスト養成読本 [ビッグデータ時代のビジネスを支えるデータ分析力が身につく! ] (Software Design plus) 」の p.38 と同様の Titanic データセットを使ったロジスティック回帰とオッズ比の算出です。
以前使ったデータは主に数値でしたが、今回は主に因子(factor)データを扱っている点が異なっています。
今回のソースは http://github.com/fits/try_samples/tree/master/blog/20140302/
はじめに
MCMCpack パッケージを R へインストールしておきます。
install.packages("MCMCpack")
Titanic データセット
Titanic のデータセットは R にデフォルトで用意されており、data.frame(Titanic) すると下記のようになります。
data.frame(Titanic) 結果
> data.frame(Titanic) Class Sex Age Survived Freq 1 1st Male Child No 0 2 2nd Male Child No 0 3 3rd Male Child No 35 4 Crew Male Child No 0 5 1st Female Child No 0 6 2nd Female Child No 0 7 3rd Female Child No 17 8 Crew Female Child No 0 9 1st Male Adult No 118 10 2nd Male Adult No 154 11 3rd Male Adult No 387 12 Crew Male Adult No 670 13 1st Female Adult No 4 14 2nd Female Adult No 13 15 3rd Female Adult No 89 16 Crew Female Adult No 3 17 1st Male Child Yes 5 18 2nd Male Child Yes 11 19 3rd Male Child Yes 13 20 Crew Male Child Yes 0 ・・・
内容は以下の通りです。
| Class(船室等級) | Sex(性別) | Age(年齢層) | Survived(生存可否) | Freq(人数) |
|---|---|---|---|---|
| 1st, 2nd, 3rd, Crew | Male, Female | Child, Adult | No, Yes | 数値 |
今回は、ロジスティック回帰の結果から船室等級・性別・年齢層毎の生存率に対するオッズ比を算出します。
(1) glm を使ったロジスティック回帰とオッズ比
書籍の内容とほとんど同じですが、
まずは glm 関数を使ったロジスティック回帰です。
Survived~. としているので Class・Sex・Age を説明変数としたロジスティック回帰を実施します。
オッズ比は exp(<推定値>) で算出できるので、書籍のような epicalc パッケージは使わず、glm 結果の $coefficients を exp 関数へ渡してオッズ比を算出しました。
logiMcmcglmm.R
d <- data.frame(Titanic) d.data <- data.frame( Class = rep(d$Class, d$Freq), Sex = rep(d$Sex, d$Freq), Age = rep(d$Age, d$Freq), Survived = rep(d$Survived, d$Freq) ) d.res <- glm(Survived~., data = d.data, family = binomial) summary(d.res) # オッズ比 exp(d.res$coefficients)
なお、上記では data.frame(Titanic) の Freq(人数) を展開したデータフレーム(下記)を使ってロジスティック回帰を実施しています。
d.data の内容
Class Sex Age Survived 1 3rd Male Child No 2 3rd Male Child No ・・・ 35 3rd Male Child No 36 3rd Female Child No ・・・
実行結果は下記の通りです。
実行結果
・・・
> d.res <- glm(Survived~., data = d.data, family = binomial)
> summary(d.res)
Call:
glm(formula = Survived ~ ., family = binomial, data = d.data)
Deviance Residuals:
Min 1Q Median 3Q Max
-2.0812 -0.7149 -0.6656 0.6858 2.1278
Coefficients:
Estimate Std. Error z value Pr(>|z|)
(Intercept) 0.6853 0.2730 2.510 0.0121 *
Class2nd -1.0181 0.1960 -5.194 2.05e-07 ***
Class3rd -1.7778 0.1716 -10.362 < 2e-16 ***
ClassCrew -0.8577 0.1573 -5.451 5.00e-08 ***
SexFemale 2.4201 0.1404 17.236 < 2e-16 ***
AgeAdult -1.0615 0.2440 -4.350 1.36e-05 ***
---
Signif. codes: 0 '***' 0.001 '**' 0.01 '*' 0.05 '.' 0.1 ' ' 1
(Dispersion parameter for binomial family taken to be 1)
Null deviance: 2769.5 on 2200 degrees of freedom
Residual deviance: 2210.1 on 2195 degrees of freedom
AIC: 2222.1
Number of Fisher Scoring iterations: 4
>
> # オッズ比
> exp(d.res$coefficients)
(Intercept) Class2nd Class3rd ClassCrew SexFemale AgeAdult
1.9844057 0.3612825 0.1690159 0.4241466 11.2465380 0.3459219
オッズ比から、女性(Female)は男性(Male)の 11倍(11.2465380)、大人(Adult)は子供(Child)の 3分の1 (0.3459219)の生存率という結果になりました。
(2) MCMClogit を使ったロジスティック回帰とオッズ比
それでは、MCMCpack の MCMClogit 関数を使って同様の処理を実施してみます。
glm 関数の場合とほとんど同じですが、MCMClogit 関数は応答変数(下記の Survived)に因子型(factor)を扱えないようなので、Survived の No と Yes を 0(= No) と 1(= Yes)へ変換しています。
(as.numeric(d$Survived) - 1 の箇所)
また、MCMClogit 関数の結果も glm とは違って値の分布となるので summary した結果の平均値(期待値) Mean を使ってオッズ比を算出しています。
logistic_odds_mcmclogit.R
library(MCMCpack) d <- data.frame(Titanic) d.data <- data.frame( Class = rep(d$Class, d$Freq), Sex = rep(d$Sex, d$Freq), Age = rep(d$Age, d$Freq), Survived = rep(as.numeric(d$Survived) - 1, d$Freq) ) d.res <- MCMClogit(Survived~., data = d.data) d.summ <- summary(d.res) d.summ #オッズ比 exp(d.summ$statistics[, "Mean"])
実行結果は下記の通りです。
実行結果
・・・
> d.res <- MCMClogit(Survived~., data = d.data)
>
> d.summ <- summary(d.res)
>
> d.summ
Iterations = 1001:11000
Thinning interval = 1
Number of chains = 1
Sample size per chain = 10000
1. Empirical mean and standard deviation for each variable,
plus standard error of the mean:
Mean SD Naive SE Time-series SE
(Intercept) 0.6775 0.2682 0.002682 0.012059
Class2nd -1.0367 0.1961 0.001961 0.008828
Class3rd -1.7935 0.1768 0.001768 0.008054
ClassCrew -0.8607 0.1596 0.001596 0.006971
SexFemale 2.4486 0.1384 0.001384 0.006279
AgeAdult -1.0540 0.2392 0.002392 0.010882
2. Quantiles for each variable:
2.5% 25% 50% 75% 97.5%
(Intercept) 0.1455 0.4992 0.6831 0.8630 1.2028
Class2nd -1.4185 -1.1683 -1.0331 -0.9027 -0.6626
Class3rd -2.1481 -1.9130 -1.7926 -1.6760 -1.4476
ClassCrew -1.1764 -0.9661 -0.8651 -0.7535 -0.5488
SexFemale 2.1543 2.3600 2.4525 2.5442 2.7110
AgeAdult -1.5275 -1.2144 -1.0573 -0.8904 -0.5907
>
> #オッズ比
> exp(d.summ$statistics[, "Mean"])
(Intercept) Class2nd Class3rd ClassCrew SexFemale AgeAdult
1.9690188 0.3546137 0.1663746 0.4228566 11.5715841 0.3485381
glm と同じような結果となりました。
最後に、それぞれの値の分布は下記のようになりました。


R で個体差のあるロジスティック回帰2 - MCMCglmm
前回 に続き、今回は個体差を考慮したロジスティック回帰を MCMCglmm で試してみます。
実は MCMCglmm の他にも MCMCpack の MCMChlogit や bayesm の rhierBinLogit 等といろいろ試そうとしてみたのですが、イマイチ使い方が分からなかったので今回は断念しました。
今回使用したサンプルソースは http://github.com/fits/try_samples/tree/master/blog/20140211/
はじめに
今回使用するパッケージを R へインストールしておきます。
install.packages("MCMCglmm")
(2) MCMCglmm を使った階層ベイズモデルによるロジスティック回帰(MCMCglmm 関数)
それでは、前回 と同じデータ (data7.csv) を使って推定と予測線のグラフ化を実施してみます。
MCMCglmm() による推定
MCMCglmm() 関数は glmmML() 関数とほぼ同じ使い方ができますが、
今回のケースでは下記の点が異なります。
- family に multinomial2 を指定する
- デフォルトで個体差 (units に該当) が考慮されるようなので特に id 列を指定する必要はない
デフォルトで詳細(処理状況)が出力されるので、下記では出力しないよう verbose = FALSE としています。
logiMcmcglmm.R
d <- read.csv('data7.csv') library(MCMCglmm) d.res <- MCMCglmm(cbind(y, N - y) ~ x, data = d, family = "multinomial2", verbose = FALSE) summary(d.res) ・・・
実行結果は下記のようになります。
実行結果
> d.res <- MCMCglmm(cbind(y, N - y) ~ x, data = d, family = "multinomial2", verbose = FALSE)
>
> summary(d.res)
Iterations = 3001:12991
Thinning interval = 10
Sample size = 1000
DIC: 667.8652
R-structure: ~units
post.mean l-95% CI u-95% CI eff.samp
units 6.988 3.898 10.58 517.4
Location effects: cbind(y, N - y) ~ x
post.mean l-95% CI u-95% CI eff.samp pMCMC
(Intercept) -4.2271 -6.3586 -2.5264 814.3 <0.001 ***
x 1.0144 0.6117 1.5237 800.3 <0.001 ***
---
Signif. codes: 0 '***' 0.001 '**' 0.01 '*' 0.05 '.' 0.1 ' ' 1
各パラメータの平均値 (post.mean) が で、前回の結果(glmmML)と近い値になりました。
ここで units は前回の個体差パラメータ の分散
に該当するようですので、標準偏差とするために平方根を取りました。
プログラム上で上記の結果を使う場合は、MCMCglmm() の結果から
(Intercept, x)の分布を
$Sol で、 (units)の分布を
$VCV で取得できます。
なお、実行する度に若干異なる値になるようなので、何らかのオプションを指定して調整する必要があるのかもしれません。
葉数と生存種子数
MCMCglmm() の結果を使って 前回 と同様にグラフ化してみます。
今回はパラメータの平均値(post.mean の値)を使わずに、事後最頻値 (posterior mode) というものを使って予測線を描画してみました。
事後最頻値は posterior.mode() 関数で取得できます。
logiMcmcglmm.R
・・・ # 生存確率の算出 calcProb <- function(x, b, r) 1.0 / (1.0 + exp(-1 * (b[1] + b[2] * x + r))) png("logiMcmcglmm_1.png") plot(d$x, d$y) xx <- seq(min(d$x), max(d$x), length = 100) # beta1 と beta2 の事後最頻値 beta <- posterior.mode(d.res$Sol) # s の事後最頻値 sd <- sqrt(posterior.mode(d.res$VCV)) lines(xx, max(d$N) * calcProb(xx, beta, 0), col="green") lines(xx, max(d$N) * calcProb(xx, beta, -1 * sd), col="blue") lines(xx, max(d$N) * calcProb(xx, beta, sd), col="blue") dev.off() ・・・
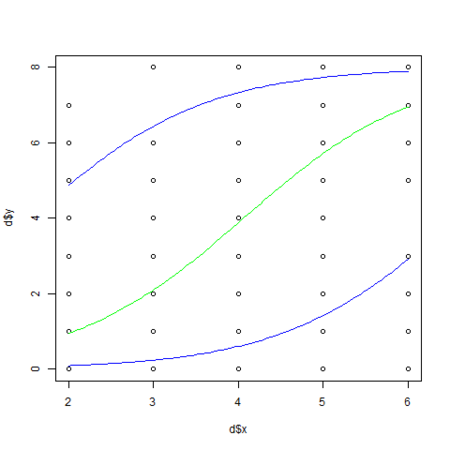
ついでに、前々回 のようにpredict() を使って予測線を描画しようとしてみましたが、今のところ predict.MCMCglmm() は新しいデータの指定に対応していないようです。
> predict(d.res, data.frame(x = xx)) Error in predict.MCMCglmm(d.res, data.frame(x = xx)) : sorry newdata not implemented yet
葉数  の種子数分布
の種子数分布
こちらも事後最頻値を使って、前回 と同様にグラフ化してみます。
logiMcmcglmm.R
・・・ png("logiMcmcglmm_2.png") yy <- 0:max(d$N) plot(yy, table(d[d$x == 4,]$y), xlab="y", ylab="num") # 葉数 x を固定した場合の生存種子数 y の確率分布を算出 calcL <- function(ylist, xfix, n, b, s) sapply(ylist, function(y) integrate( f = function(r) dbinom(y, n, calcProb(xfix, b, r)) * dnorm(r, 0, s), lower = s * -10, upper = s * 10 )$value ) lines(yy, calcL(yy, 4, max(d$N), beta, sd) * length(d[d$x == 4,]$y), col="red", type="b") dev.off()
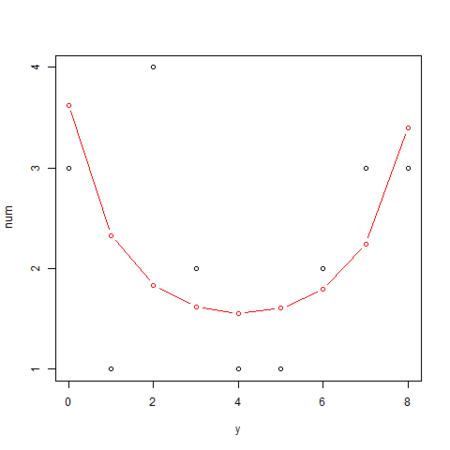
R で個体差のあるロジスティック回帰1 - glmmML
前回 のロジスティック回帰に続き、書籍 「 データ解析のための統計モデリング入門――一般化線形モデル・階層ベイズモデル・MCMC (確率と情報の科学) 」のサンプルを使って個体差を考慮したロジスティック回帰を GLMM と階層ベイズモデルで試してみます。
- (1) GLMM によるロジスティック回帰 (glmmML 関数)
- (2) MCMCglmm を使った階層ベイズモデルによるロジスティック回帰(MCMCglmm 関数)
ただし、今回は (1) だけで (2) に関しては次回に書く予定です。
サンプルソースは http://github.com/fits/try_samples/tree/master/blog/20140209/
はじめに
パッケージのインストール
今回使用するパッケージを R へインストールしておきます。
install.packages("glmmML")
データ
データは書籍のサポート web サイトから取得します。 今回は 7章「一般化線形混合モデル(GLMM)- 個体差のモデリング -」のサンプルデータを使います。
データ data7.csv
"N","y","x","id" 8,0,2,1 8,1,2,2 8,2,2,3 ・・・
データ内容は下記の通りです。
| 項目 | 内容 |
|---|---|
| N | 調査種子数 |
| y | 生存種子数 |
| x | 植物の葉数 |
| id | 個体のID |
葉数 の値が 2 ~ 6 までとなっており、
葉数毎に 20 個体ずつの生存種子数
をサンプリングしたデータとなっています。 (合計 100 個体)
二項分布の性質と過分散
ところで、二項分布の性質として下記があります。
今回のケースでは n が調査種子数(= 8)、p が生存確率です。
- (1) 平均(期待値とするのが適切かも)が
- (2) 分散が
ここで、今回のデータは下記のようになっています。
葉数 4(x = 4)場合の平均と分散
> mean(d[d$x == 4,]$y) [1] 4.05 > var(d[d$x == 4,]$y) [1] 8.365789
np = 4.05 とすると p = 4.05 / 8 = 約 0.5 となり、二項分布として期待される分散は np(1 - p) = 8 × 0.5 × (1 - 0.5) = 2 で、実際の分散 8.36 の方が明らかに大きい値となっています。 (過分散)
二項分布として考えると、原因不明な個体差等による効果を考慮した統計モデルを使ったロジスティック回帰が必要となり、GLMM や階層ベイズを使用するという流れとなるようです。
(1) GLMM によるロジスティック回帰 (glmmML 関数)
それでは glmmML() 関数を使った推定値の算出とグラフ化を実施してみます。
推定方法
個体差を考慮するため、GLMM では個体差のパラメータ を追加した線形予測子
を使う事になるようです。
ここで、 は生存確率です。
そして、 が正規分布の確率分布に従っていると仮定し、
確率密度関数 を加味して
を積分した
尤度 に対して
全データの積 の対数尤度
が最大になるようなパラメータ
の最尤推定値を求めます。
なお、 は二項分布の確率密度関数で、
は個体差
のばらつき(標準偏差)です。
glmmML() による推定
glmmML() 関数へ指定するオプションは、前回 の glm() とほとんど同じですが 、下記の点が異なります。
- 個体差のパラメータ
部分を cluster オプションで指定
今回は個体毎に異なる値が設定されている id 列を cluster オプションへ指定します。
logiGlmmML.R
d <- read.csv('data7.csv') library(glmmML) d.res <- glmmML(cbind(y, N - y) ~ x, data = d, family = binomial, cluster = id) summary(d.res) ・・・
glmmML() による推定結果は下記の通りです。
実行結果
Call: glmmML(formula = cbind(y, N - y) ~ x, family = binomial, data = d, cluster = id)
coef se(coef) z Pr(>|z|)
(Intercept) -4.190 0.8777 -4.774 1.81e-06
x 1.005 0.2075 4.843 1.28e-06
Scale parameter in mixing distribution: 2.408 gaussian
Std. Error: 0.2202
LR p-value for H_0: sigma = 0: 2.136e-55
Residual deviance: 269.4 on 97 degrees of freedom AIC: 275.4
と推定されました。
なお、glmmML() の結果から、
の値は
$coefficients で、 の値は
$sigma で取得できます。
葉数と生存種子数
推定したパラメータを使って葉数 x と生存種子数 y の予測線をグラフ化する処理は下記のようになります。
生存種子数の予測値は 調査種子数(= max(d$N) = 8)× 生存確率 で算出し、
生存確率 は
の式を元に算出します。
logiGlmmML.R
・・・ # 生存確率の算出 calcProb <- function(x, b, r) 1.0 / (1.0 + exp(-1 * (b[1] + b[2] * x + r))) png("logiGlmmML_1.png") # x と y をプロット plot(d$x, d$y) # x の範囲(2 ~ 6)を 100分割 xx <- seq(min(d$x), max(d$x), length = 100) beta <- d.res$coefficients # 個体差 r = 0 の葉数と生存種子数との予測線 lines(xx, max(d$N) * calcProb(xx, beta, 0), col="green") # 個体差 r = -2.408 の葉数と生存種子数との予測線 lines(xx, max(d$N) * calcProb(xx, beta, -1 * d.res$sigma), col="blue") # 個体差 r = 2.408 の葉数と生存種子数との予測線 lines(xx, max(d$N) * calcProb(xx, beta, d.res$sigma), col="blue") dev.off() ・・・
個体差 r = 0 (個体差なし)とした場合の予測線を緑色で、 r = ±2.408 の予測線を青色で描画しています。

葉数  の生存種子数分布
の生存種子数分布
次に、葉数 x が 4 の場合の生存種子数 y と個体数の分布と予測線をグラフ化する処理は下記のようになります。
glmmML() の結果から、x = 4 の場合の y の確率分布を算出し、x = 4 のサンプル数(= 20)を乗算して予測線を描画しています。
y の確率分布は、0 ~ 8 のそれぞれの値に対して で算出します。 (二項分布 × 正規分布 を個体差
で積分)
下記では sapply() と integrate() 関数を使って上記の算出処理を実装しています。
なお、lower と upper オプションを使って積分範囲を から
としています。
logiGlmmML.R
・・・ png("logiGlmmML_2.png") # 0 ~ 8 yy <- 0:max(d$N) # 生存種子数 y と個体数の分布 plot(yy, table(d[d$x == 4,]$y), xlab="y", ylab="num") # 葉数 x を固定した場合の生存種子数 y の確率分布を算出 calcL <- function(ylist, xfix, n, b, s) sapply(ylist, function(y) integrate( f = function(r) dbinom(y, n, calcProb(xfix, b, r)) * dnorm(r, 0, s), lower = s * -10, upper = s * 10 )$value ) # 予測線 lines(yy, calcL(yy, 4, max(d$N), beta, d.res$sigma) * length(d[d$x == 4,]$y), col="red", type="b") dev.off()

今回はここまで。 MCMCglmm を使った階層ベイズは次回。
R でロジスティック回帰 - glm, MCMCpack
前回 に続き、今回も書籍 「 データ解析のための統計モデリング入門――一般化線形モデル・階層ベイズモデル・MCMC (確率と情報の科学) 」のサンプルを使って GLM とベイズ統計を試してみます。
題材は、6章「GLM の応用範囲をひろげる -ロジスティック回帰など-」のサンプルデータを使ったロジスティック回帰です。
- (1) GLM によるロジスティック回帰 (glm 関数)
- (2) MCMCpack を使ったベイズ統計によるロジスティック回帰(MCMCmetrop1R 関数)
ここで、MCMCpack のロジスティック回帰に MCMClogit 関数を使えると思ったのですが、使い方がよく分からなかったので MCMCmetrop1R 関数のみ利用しました。
サンプルソースは http://github.com/fits/try_samples/tree/master/blog/20131229/
はじめに
MCMCpack パッケージを install.packages("MCMCpack") で R の実行環境へインストールしておきます。
ロジスティック回帰を試すためのデータは書籍のサポート web サイトから取得できます。
データ data4a.csv
N,y,x,f 8,1,9.76,C 8,6,10.48,C 8,5,10.83,C ・・・
データ内容は下記のようになっており、個体 i それぞれにおいて 「 個の観察種子のうち生きていて発芽能力があるものは
個、死んだ種子は
個」 となっています。
| 項目 | 内容 |
|---|---|
| N | 観察種子数 |
| y | 生存種子数 |
| x | 植物の体サイズ |
| f | 施肥処理 (C: 肥料なし, T: 肥料あり) |
体サイズ x と肥料による施肥処理 f が種子の生存する確率(ある個体 i から得られた 1個の種子が生存している確率)にどのように影響しているかをロジスティック回帰で解析します。
(1) GLM によるロジスティック回帰 (glm 関数)
まず、glm 関数を使ったロジスティック回帰を行って予測値の曲線を描画します。
特徴は以下のようになります。
- family に binomial (二項分布)を指定 (デフォルトのリンク関数が logit となる)
- family が binomial で応答変数 (
cbind(y, N - y)の箇所) が 2値(0, 1)以外なら cbind する必要あり (2値なら y だけでよい)
下記では、生存する確率 の関数が
、
線形予測子 で解析する事になります。
logisticGlm.R
d <- read.csv('data4a.csv') d.res <- glm(cbind(y, N - y) ~ x + f, data = d, family = binomial) summary(d.res) png("logisticGlm.png") # C: 肥料なしを 赤、T: 肥料ありを 青 で描画 plot(d$x, d$y, col = c("red", "blue")[d$f]) xx <- seq(min(d$x), max(d$x), length = 1000) ft <- factor("T", levels = levels(d$f)) fc <- factor("C", levels = levels(d$f)) # 下記でも可 #ft <- factor("T", levels = c("C", "T")) #fc <- factor("C", levels = c("C", "T")) # T: 肥料ありの予測値を算出 qq.t <- predict(d.res, data.frame(x = xx, f = ft), type="response") # C: 肥料なしの予測値を算出 qq.c <- predict(d.res, data.frame(x = xx, f = fc), type="response") # T: 肥料ありの予測値の曲線を 緑 で描画 lines(xx, max(d$N) * qq.t, col = "green") # C: 肥料なしの予測値の曲線を 黄 で描画 lines(xx, max(d$N) * qq.c, col = "yellow") dev.off()
predict 関数を使えば glm 結果の回帰モデルから新しいデータによる予測値を求めることができますので、"T:肥料あり" と "C:肥料なし" のそれぞれの予測値を求めて曲線を描画しています。
predict の結果は、生存する確率 の予測値ですので、縦軸が生存種子数
のグラフへ描画するには観察種子数 N (ここでは 8)を乗算する事になります。
ここで、"T:肥料あり" と "C:肥料なし" は factor 関数を使ってファクタ(因子)として作成しています。
d$f と同じ水準を持たせる必要がありますので levels 指定しています。
また、predict には glm で使った説明変数と同じ名前(上記の x と f)を使ったデータを渡す点に注意が必要です。
実行
実行すると下記のような結果になりました。
> R CMD BATCH logisticGlm.R
実行結果 logisticGlm.Rout
・・・
> d.res <- glm(cbind(y, N - y) ~ x + f, data = d, family = binomial)
>
> summary(d.res)
Call:
glm(formula = cbind(y, N - y) ~ x + f, family = binomial, data = d)
Deviance Residuals:
Min 1Q Median 3Q Max
-3.2584 -0.7948 0.1753 0.8757 3.1589
Coefficients:
Estimate Std. Error z value Pr(>|z|)
(Intercept) -19.5361 1.4138 -13.82 <2e-16 ***
x 1.9524 0.1389 14.06 <2e-16 ***
fT 2.0215 0.2313 8.74 <2e-16 ***
---
Signif. codes: 0 '***' 0.001 '**' 0.01 '*' 0.05 '.' 0.1 ' ' 1
(Dispersion parameter for binomial family taken to be 1)
Null deviance: 499.23 on 99 degrees of freedom
Residual deviance: 123.03 on 97 degrees of freedom
AIC: 272.21
Number of Fisher Scoring iterations: 5
・・・
この結果より、線形予測子 のパラメータ推定値
が
-19.5361, 1.9524, 2.0215 となります。

(2) MCMCpack を使ったベイズ統計によるロジスティック回帰(MCMCmetrop1R 関数)
次は、MCMCpack の MCMCmetrop1R 関数を使ったロジスティック回帰です。
今回のモデルの対数尤度は で、
、
なので、
これらを関数化 (下記の func) して MCMCmetrop1R 関数へ渡しています。
ちなみに、 です。(combination)
前回は、項目毎のデータ(d$x や d$y)を func 関数へ渡すようにしていましたが、今回はデータ d を丸ごと渡すようにしました。 MCMCmetrop1R(・・・, data = d, ・・・)
ファクタ(因子)のデータ (d$f) を直接計算に使えないようなので as.numeric で数値化して使っています。
なお、 の部分は
dbinom と log 関数を使うとシンプルになります。
パラメータ
の初期値は
theta.init で c(0, 0, 0) と指定しました。
logisticMcmcMetrop.R
library(MCMCpack) d <- read.csv('data4a.csv') func <- function(beta, data) { z <- beta[1] + beta[2] * data$x + beta[3] * as.numeric(data$f) q <- 1.0 / (1.0 + exp(-z)) sum(log(choose(data$N, data$y)) + data$y * log(q) + (data$N - data$y) * log(1 - q)) # 下記でも可 # sum(log(dbinom(data$y, data$N, q))) } d.res <- MCMCmetrop1R(func, theta.init = c(0, 0, 0), data = d, logfun = TRUE) summary(d.res) png("logisticMcmcMetrop.png") plot(d$x, d$y, col = c("red", "blue")[d$f]) xx <- seq(min(d$x), max(d$x), length = 1000) ft <- factor("T", levels = levels(d$f)) fc <- factor("C", levels = levels(d$f)) zt <- mean(d.res[,1]) + mean(d.res[,2]) * xx + mean(d.res[,3]) * as.numeric(ft) zc <- mean(d.res[,1]) + mean(d.res[,2]) * xx + mean(d.res[,3]) * as.numeric(fc) lines(xx, max(d$N) * 1.0 / (1.0 + exp(-zt)), col="green") lines(xx, max(d$N) * 1.0 / (1.0 + exp(-zc)), col="yellow") dev.off()
MCMCmetrop1R 結果の各パラメータの mean の値を使って、
肥料の有無(T と C)の生存確率の予測値をそれぞれ算出して、
観察種子数 N (ここでは 8)を乗算し曲線を描画しています。
実行
実行すると下記のような結果になります。
> R CMD BATCH logisticMcmcMetrop.R
実行結果 logisticMcmcMetrop.Rout
・・・
@@@@@@@@@@@@@@@@@@@@@@@@@@@@@@@@@@@@@@@@@@@@@@@@@@@@@@@@@
The Metropolis acceptance rate was 0.44590
@@@@@@@@@@@@@@@@@@@@@@@@@@@@@@@@@@@@@@@@@@@@@@@@@@@@@@@@@
>
> summary(d.res)
Iterations = 501:20500
Thinning interval = 1
Number of chains = 1
Sample size per chain = 20000
1. Empirical mean and standard deviation for each variable,
plus standard error of the mean:
Mean SD Naive SE Time-series SE
[1,] -21.694 1.5583 0.0110185 0.037186
[2,] 1.966 0.1386 0.0009798 0.003322
[3,] 2.027 0.2268 0.0016035 0.005383
2. Quantiles for each variable:
2.5% 25% 50% 75% 97.5%
var1 -24.836 -22.733 -21.667 -20.596 -18.766
var2 1.703 1.870 1.963 2.059 2.248
var3 1.594 1.873 2.022 2.172 2.494
・・・
Mean の値が glm の結果と似たような値になりました。 グラフにすると違いが分からないくらいです。

 の分布
の分布

 の分布
の分布

 の分布
の分布
